Bakgrund
Måste överkomma en aktiveringsenergi, staven måste vara tillräckligt böjd för att kunna knäckas.
De flesta reaktioner kräver en slags aktiveringsenergi.
Det man vill åstakomma är utbyte av valenselektroner → skapa nya bindingarna
I vätskor sker det slumpvis, rör och knuffar på sig. För att de ska komma tillräckligt nära och byta ut valenselektroner måste prova många olika knuffar för att få rätt
Höja sannolikoheten att de träffas på rätt sätt och rät tkonfiguration
En bildning av ett enzymkomplex som sänker den här aktiveringsenergin, de underlättar för det här övergångstillståendet att bildas.
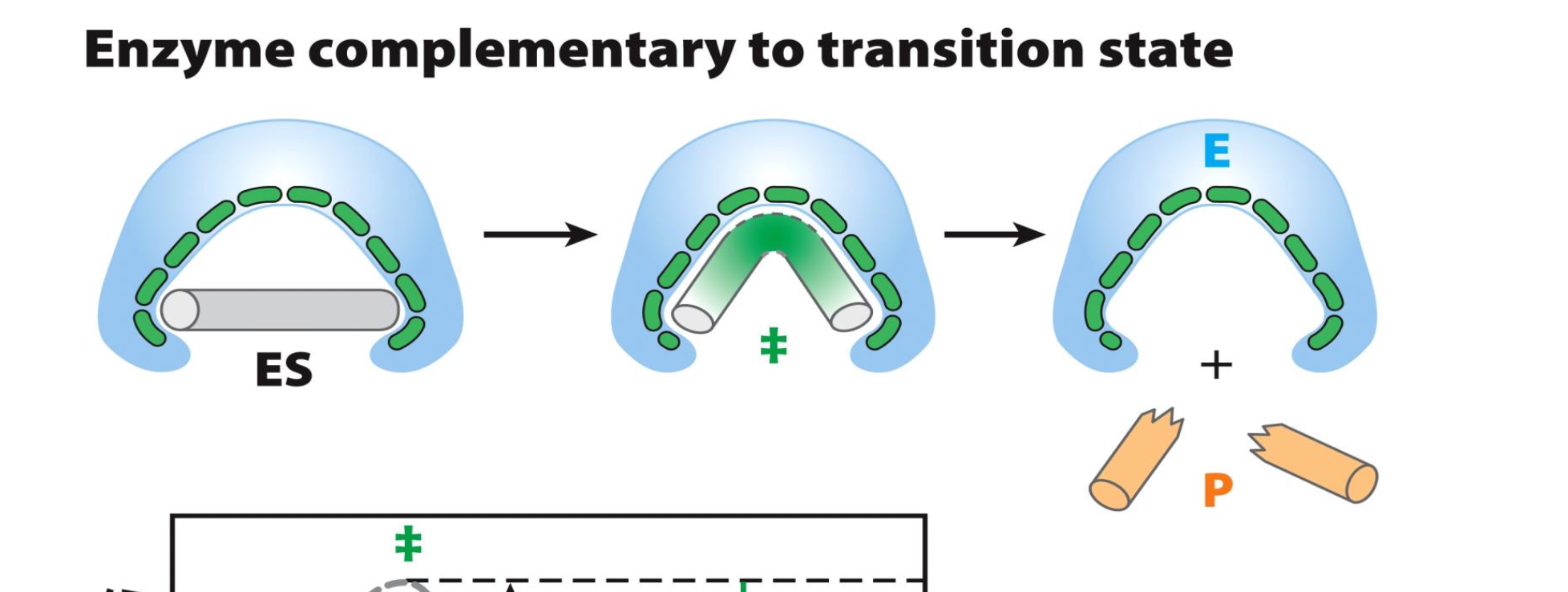
Gillar inte den modellen för att de finns inte magneter i alla enzymer, det är bara slumpen som bestämmer hur och om en substrat inte agerar med sitt enzym, det är en knuffa och slumpmässiga interaktioner som bestämmer om substratet hittar ett enzym, ingen dragsningskraft i det, utöver det är det en bra tankemodel, att det kan tvinga substratet för att påskynda reaktionen, det är det viktigasete. Enzymerna gör det med en med en katalytisk klyfta, den är särskiljt uppbyggt för att uppnås selektivtet ett snäft, molekyler som passar just lär, en unik kemisk miljö för att uppnå TS, sluter ut vattenmolekylerna som ska utesluta eftersom de inte behövs
Hur snabbt kan ett enzym arbeta?
Michaelis-Menten
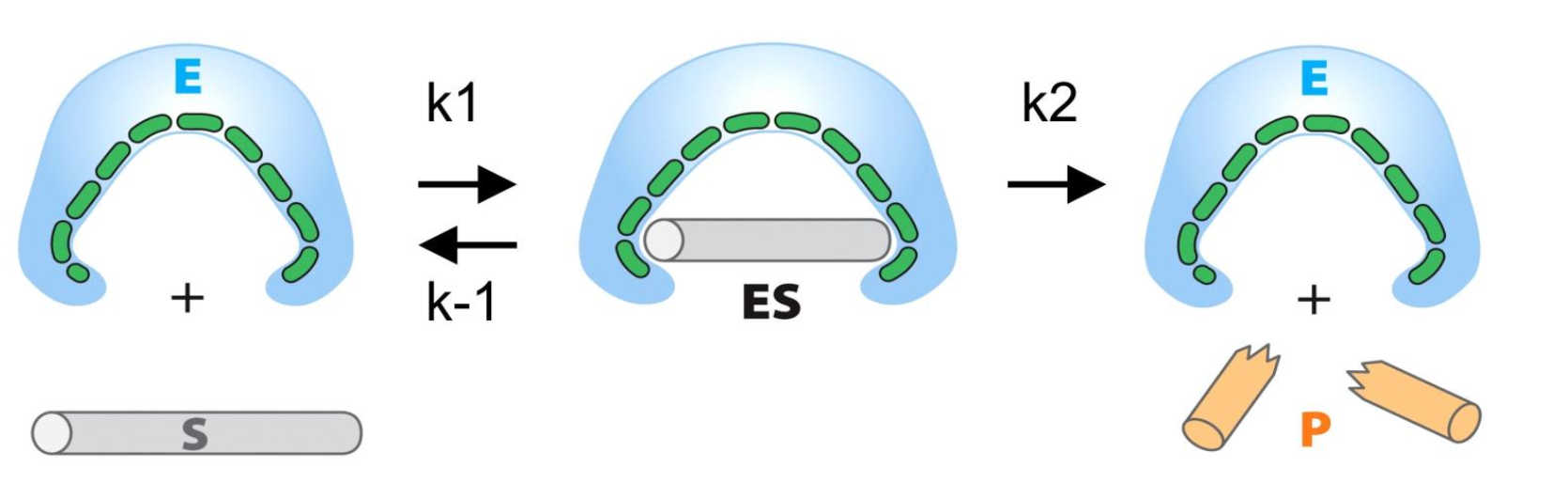
| Konstant | Beskrivning |
|---|---|
| E | enzym |
| S | substrat |
| P | produkt |
| k1 | associationskonstant ES komplex |
| k - 1 | dissociationskonstant ES komplex |
| k2 | associationskonstant produktfrisättning |
| Hur sannolikt är det att en klyfta agerar, ofta slumpmässigt kontrollerat med hjälp av diffusion, men klyfta på rätt sätt | |
| Förutsättningar: |
- interaktion som är enkelt som inte kräver stora förändringar i enzymet
- försumbart att titta på den omvända reaktionen,
- betrakta reaktionen i inledningsfasen, kan försummas
- Koncentrationen av ES är konstant (steady-state)
- dvs det finns en stabil mängd substrat, det förändras inte
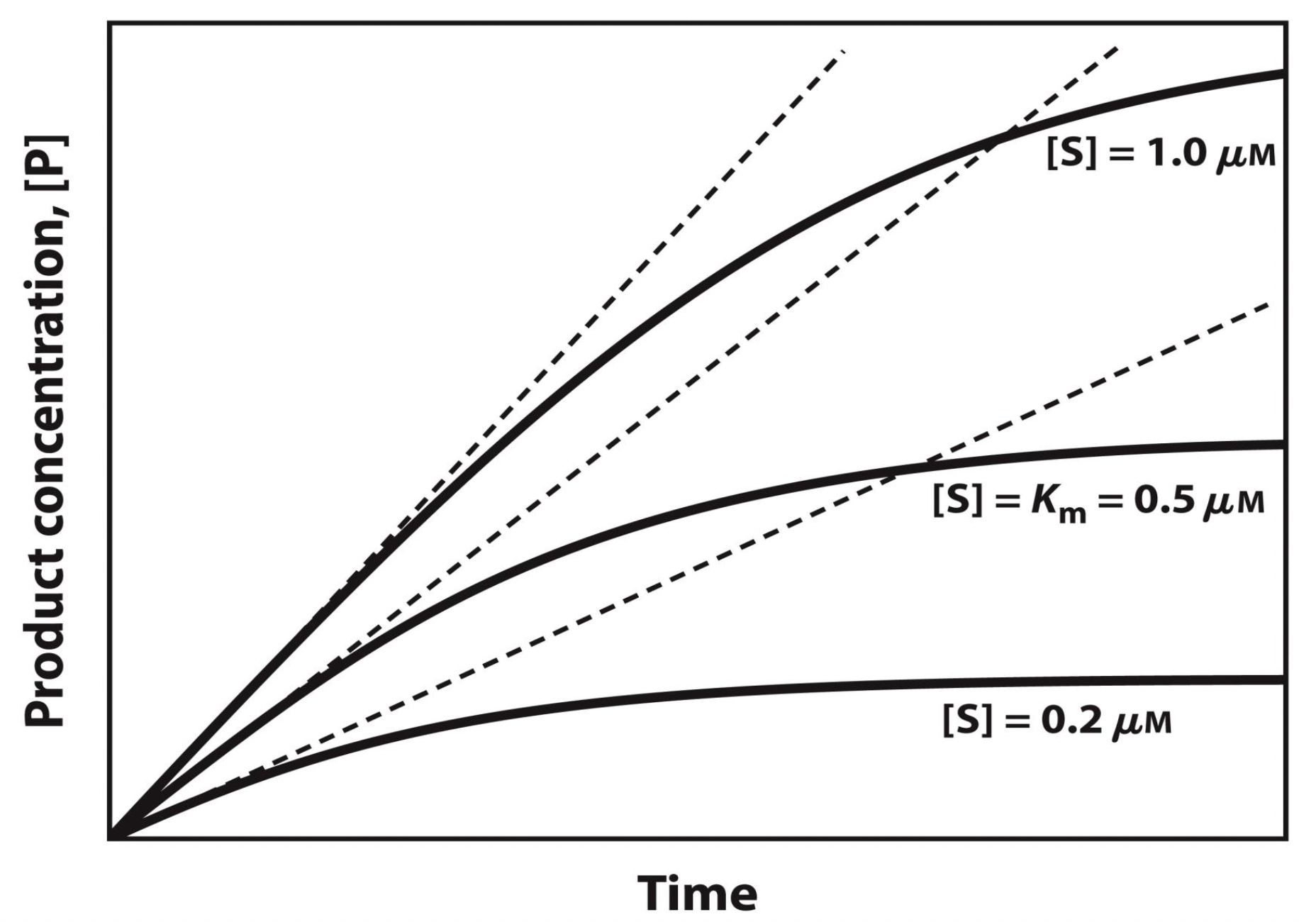 Streckade linjen är den ursprungliga lutningen på grafen
Med högre mängd substrat så får man en inledande större lutningen, mindre stigning ju mindre substrat man ger. Det tyder att det tar längre tid för substratet att binda till proteinet.
Streckade linjen är den ursprungliga lutningen på grafen
Med högre mängd substrat så får man en inledande större lutningen, mindre stigning ju mindre substrat man ger. Det tyder att det tar längre tid för substratet att binda till proteinet.
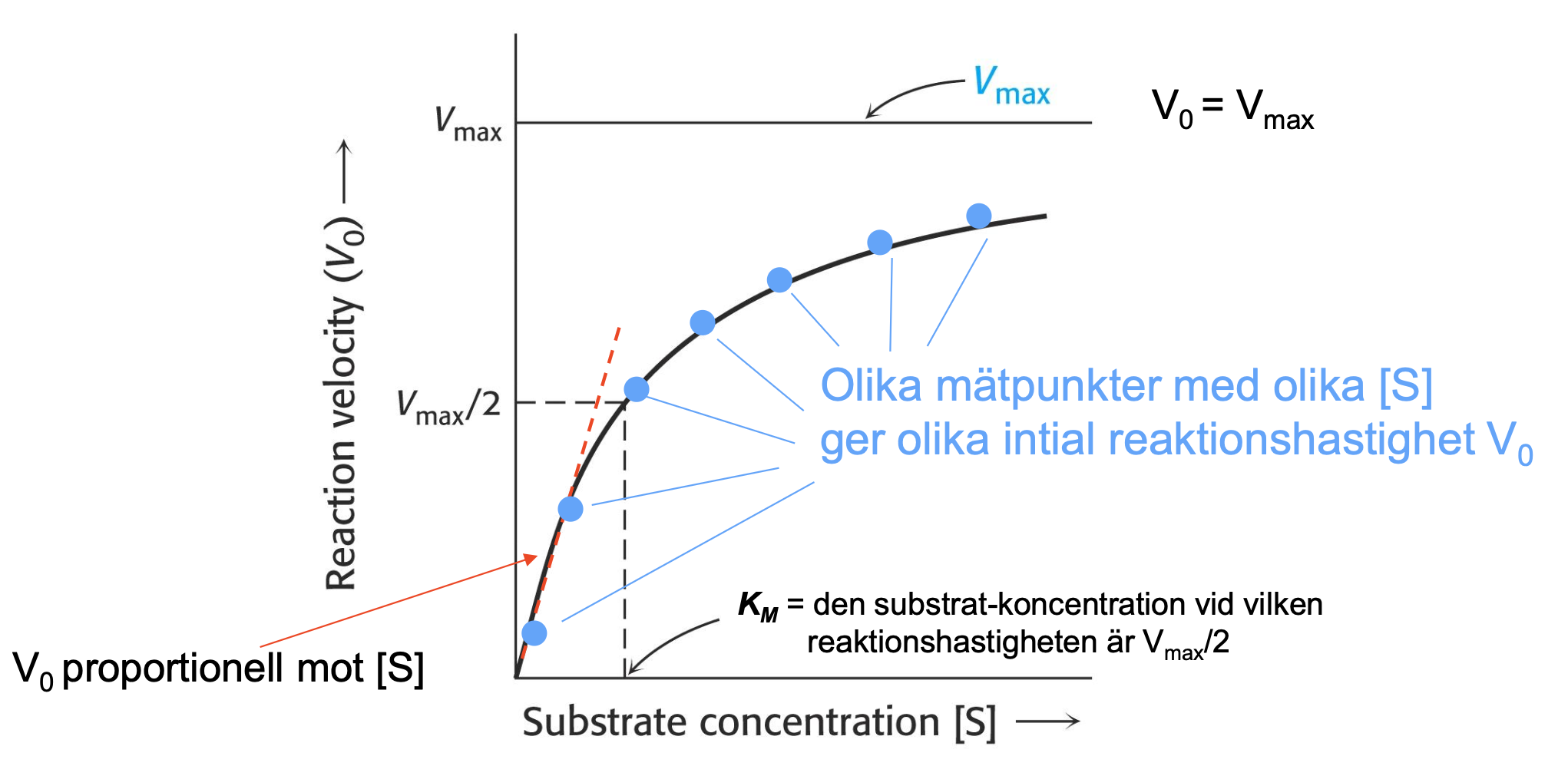 Man kan se:
Man kan se:
- Maximala reaktionshastigheten är den kan bara nå en viss hastighet, går inte att gå över.
- Man kan se hur kurvan ser ut vid mindre substratkoncentrationr
- Vid halva maximala reaktionshastigheten finns en definerad konstratkoncentration vid halva vmax, den säger hur pass sannolikt det är att bilda en stabil substratkomplex
- vid vmax är substratkoncentrationen så högt att varje moment en enzym är ledig, för den har just frisatt produkt är den bunden med ett nytt substrat, då kan man nå den totala reaktionshastigheten
- vmax/2
- halva är katalyserad
- halva är tillgänlig
- Specifik för varje enzym
Kallas för Michaelis-Mentes konstant
- Michaelis-Mentens konstant definieras (under de givna antagandena) som den när
- [S] = KM så är [E] = [ES], dvs hälften av alla enzymer är bundna i ES-komplex
- Lågt betyder stabilt ES
**Maximala reaktionshastigheten,
När substratkoncentrationen är så hög att alla enzymmolekyler är bundna i ES-komplex är reaktionshastigheten maximal, Vmax.
Antalet moleklyer P som en E kan bilda per sekund, kallas turnover number (omsättningstal)
Diffusionskontrollerad hur sannoligt är det att de slumpmässigt, när de reagerar reagerar de direkt. Kan ske upp till 600 000 ggr per sekund, medans DNA polymeras kan bara köra 15ggr per sekund eftersom den måste vara väldigt noggrand och kontrollera att allt blir rätt
Man mäter KM och VMax genom att ta flera punkter med olika sbustratkoncentrationer, ju lägre KM desto bättre kan den hålla fast, desto sämre är affiniten. Affiniteten är viktig, den säger under vilka konstellation kan man effiktivt etablera ett substratkomplex och bestämma reaktionsahastighet.
Allosterea enzymer som hemoglobin som har kooperativitet följer inte michaelis-menten-kinetik de andra subenheterna kan förändras och då krävs en annan sorts mekanism i dessa enzym för att uppnå full reaktionshastighet.
Hur kan man hämma ett enzym?
Inhibitorer har på enzymer De har sin egna affinitet, ju högre affinitet, ju bättre och mindre molekyler måste man ta in
Man analyserar affiniteten och
- irreversibala inhibitorer - binder mycket hårt
- reversibla inhibitorer - binder och släpper
E + I ⇌ EI
Competitive inhibiter
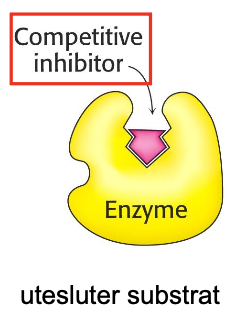 Den vanliga substraten kan inte sitta eftersom den konkurrar ut substratet
Den vanliga substraten kan inte sitta eftersom den konkurrar ut substratet
För att reaktionen ska kunna ske med en inhibitor måste man öka substratet så det har en chans.
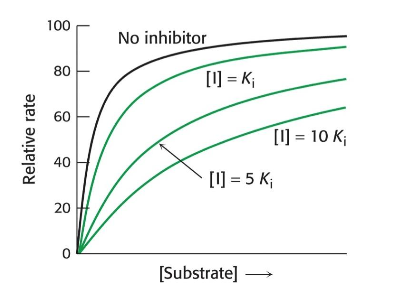 Svarta går ganska svart
När man sätter till I, så flakar den av, men den går mot samma reaktionshastighet med tillräckligt stora mängder substrat. Varje gång en enzym är ledig tillsätter den en.
Ju starkar inhibitor ju hägre K värde, KM blir större och större och affiniteten ökar.
Svarta går ganska svart
När man sätter till I, så flakar den av, men den går mot samma reaktionshastighet med tillräckligt stora mängder substrat. Varje gång en enzym är ledig tillsätter den en.
Ju starkar inhibitor ju hägre K värde, KM blir större och större och affiniteten ökar.
Non-competitive inhibitor
Binder på ett annat ställe men ändrar konformationsformen, ingen katalys kan ske.
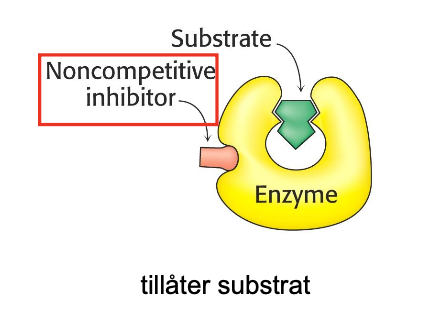 Nu finns det två olika tillstånd för varje enzym.
Nu finns det två olika tillstånd för varje enzym.
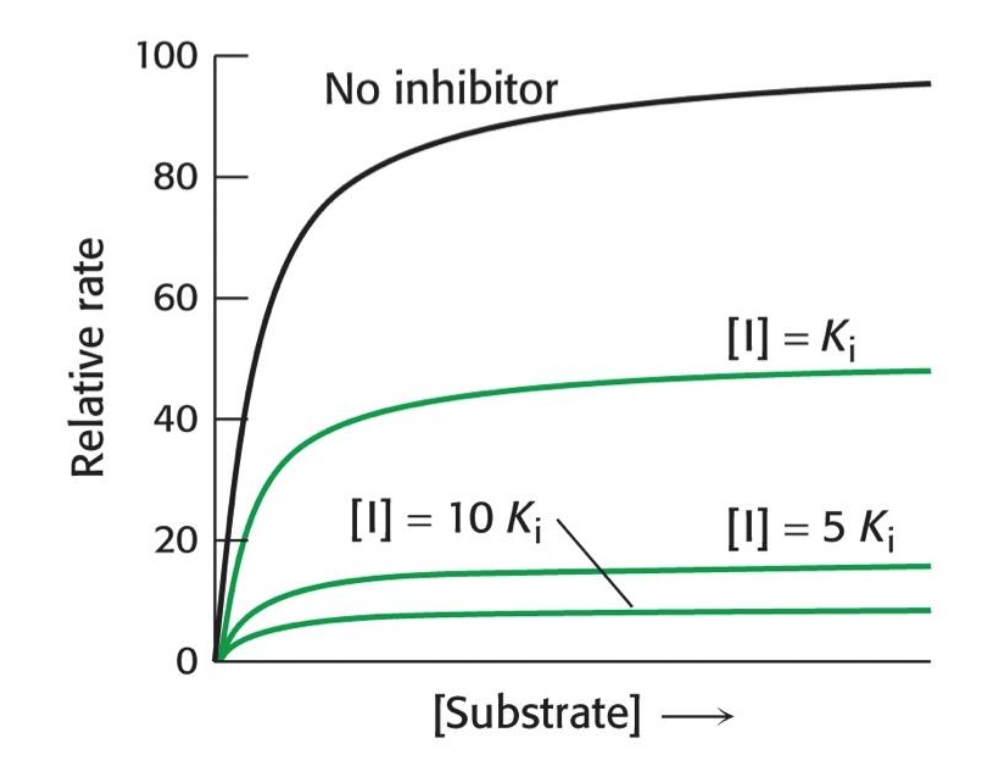 Kinetiska effekten är att man slår ut ett antal enzymer som inte är tillgängliga för katalyz, då sänker man mätbara eftersom några är utslagna och inte kan göra någonting, kan fortfarande översätta substrat till produkt. Man får samma affinitet den här reaktionen. Man slår ut enzymerna, man reducerar totalt antal tillgängliga.
Kinetiska effekten är att man slår ut ett antal enzymer som inte är tillgängliga för katalyz, då sänker man mätbara eftersom några är utslagna och inte kan göra någonting, kan fortfarande översätta substrat till produkt. Man får samma affinitet den här reaktionen. Man slår ut enzymerna, man reducerar totalt antal tillgängliga.
Inhibitorn kan kossa, den har sin egen affinitet? Hur ser man det i grafen.
Varför vill man göra det?
Många läkemedel är riktigade till det aktiva sätet för enzymerna. Aktiva centrumet binder översgångstillståndet bättre än substratet.
Exempel penicillin:
- Penicillin åstadkommer irreversibel inhibition av enzymet glykopeptid-transpeptidas, som bygger upp bakteriers cellväggar.
- Kan bygga till det aktiva sätet på glykopeptid-transpeptidas, man skapar en bindning
- den här kovalenta bindingen kan enzymet inte får bort, en kovalent binding enzym komplex kan man slå ut med hög efektivitet och bakterinen kan inte bygga upp en cellvägg
Proteolys
Hydrolys av peptidbindning Koka sojabönor
Väldigt långsamt, kräver spjälkning, 10-1000 år halveringstid I kroppen går det milliskunder-tidskalar enzymer som katalyserar proteolys = proteaser
Chymotrypsin
Ett serin proteas substrat: peptid-bindning efter Phe/Met
Den klassiska triaden (Ser-His-Asp):
- Aspartat negativt laddad, stabiliserar histidin och gör att hela systemet hålls i rätt laddningsfördelning.
- Histidin fungerar som en proton-shuttle och gör serin mycket mer reaktiv.
- Serin har en alkoholgrupp fungerar som den nukleofila attackpunkten.
- Histidin drar bort protonen från serins OH-grupp
väldigt snabb, 200ggr per sekund
- Asp His Ser
- Asp drar åt sen His, His drar åt protonen från Ser
- nu är syre atomkärnan aktiverad och väldigt reaktiv
- När man placerade en peptidbindning ovanpå det aktiva sätet kan syreatomen från Ser attackera kol-atomen
- När attacken har lyckats skapar man ett kortlivat övergångstillstånd skapar ett kol 4.5 bindingar
- Fallar ihop till en konfiguration med en ny bildning mellan kol och syre, nu är peptidbindningen spjälkad mellan kväve och kol
- nu kan ena hälften gå
- andra hälften är fortfarande bunden till serin-resten
Reaktion 2 är samma sak, men använder nu vatten för att få bort bindingen
- börjar med att peptidresten är bunden med syret i Serin
- nu kommer vatten in som en reaktant i den här reaktionen, vatten placeras in brevid histidin, alfa kolet drar åt sig en proton från ett annat ställe
- Histidin drar åt sig en proton, då blottas protonerna från en mvatten moelkyl, syre atomen blir aktiverad och mkt reaktiv, och kan igen aktivera bindingen
- skapar en kortlivad intermediär, som faller isär och bildar ett tillstånd, dragen till här histidin med en proton som kommer ifrån vatten, den andra resterade delen är bunden till andra hälften av peptid
- sen är enzymet återställt och kan katalysera en ny peptidbindningen
Detta enzymet gör att man kan designa en kemisk reaktion, men med en mkt högre specifitet från att man binder till det aktiverade syreatomen precis ovanpå sitt substrat. VIKTIGT: skapar med en sån peptidkatalyseri en konformation mellan reaktanterna som hjälper till med katalysen VIKTIGT: man har nu etablerat ett alternativ till det vanliga katalysvägen, en enzymkatalyserat man skapar specifitet genom att ta in reaktanterna, skapa en kemisk mijö som är gynnsam, mer reaktiv
Oxyanion hole är en del av enzymet för att stabilisera det instabila övergångstillståndet, som bara har en enkelbinding, drar åt sig elektronerna från två andra säten. Kol har fortfarande 4 bindingar men görs instabil. dubbelbinding till enkelbinding
Viktig aspekt, Hydrofob ficka ger ett substrat specificitet. Genom att ändra fickan så ändrar man substratets specificitet.
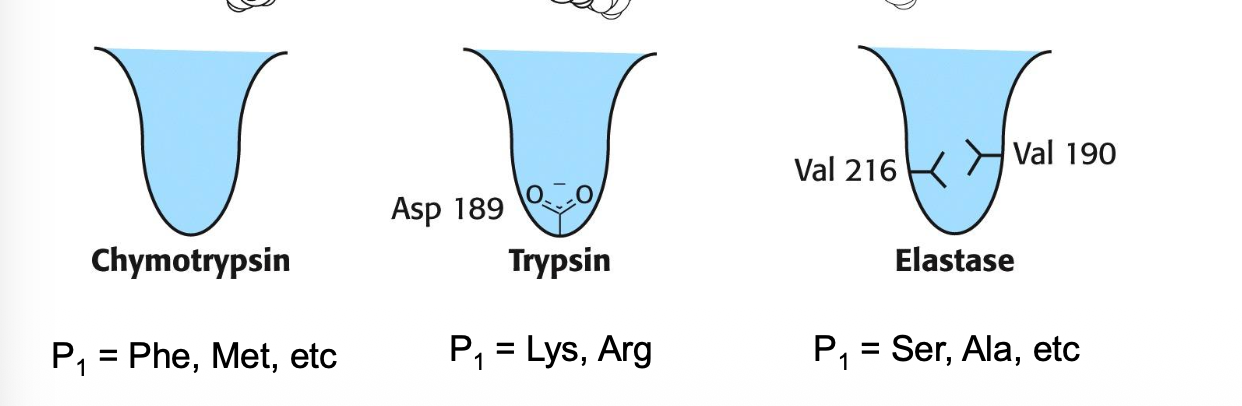
Chymotrypsin: mindre specifik, hydrofob Trypsin: hydrofob och kan etablera jonbinding med Lysin eller Arginin Elastas: här kan man binda in serin/alanin, fickan är mindre, två valin
Proteaser
Cysteinprotas Aspartylproteas Metalloproteas
Proteaser är en viktig klass av proteiner, mål för mycket läkemedel. HIV-virus, finns en proteas som spjälkar en polypeptidkedja i viruskapsylen, man har läkemedel som förstår proteasen, om den är förstörd kan man inte bygga nya partiklar.
Oxidation och reduktion (redox)
Förändrar molekylernas bindningar genom att ta in eller föra bort elektroner. Då använder man av oxyreduktaser De behöver en kofaktor för att föra över elektronerna till/från medelet. Exempel:
- etanol (oxidation)
- acetaldehyd (reduktion)
- NAD+ är en vanlig elektronbärare
- NAD+ lägger till två elektroner och en H+
Andningskedjan kontrolleras av oxidoreduktaser.
Syntaser/Ligaser
Bildar nya bindingar mellan biomolekyler
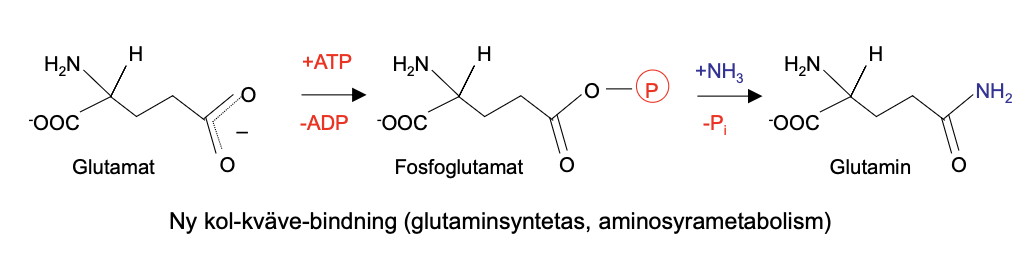 För att skapa Glutamin krävs energi för att skapa den via ATP.
Efter fosfylesering med en ny energirikbindning som man sen kan byta ut mot en ny bindning.
För att skapa Glutamin krävs energi för att skapa den via ATP.
Efter fosfylesering med en ny energirikbindning som man sen kan byta ut mot en ny bindning.
DNA-polymeras kopierar DNA genom att skapa nya fosfodiesterbindningar.
Kinaser
Använder ATP som substrat och för över en fosfatrest från ATP till en annan molekyl
Fosfataser tar bort ATP-gruppen, tillsammans med kinaser reglerar man en process genom aktiverar eller desaktiverar.
Sammanfattning
Enzymkinetik: (K=visst substrat, affinitet) och (maximala reaktionshastigheten) Enzyminhibitorer: olika typer vilken kinetisk effekt de tar
- tävlar
- allostera Enzymer:
- proteaser
- chymotrypsin detaljerat
- många år → flera gånger per sekund via katalysator
- oxidoreduktaser
- ligaser
- kinaser/fosfataser