Claes Gustavsson Biomedicin Genexpression, DNA-replikation, kommer tillbaka på T3
Basala mekanismer vid transkription
Bakterier kommer upp på föreläsningen Prokaryota:
- Har inget kärnmembran
- Många av dessa processer sker på samma plats Eukaryota
- Mer tillfälle för reglering, en viktig aspekt,
- Det är så vi styr vilka proteiner som ska finnas i en given cell
- Många olika processer, kön/fett/lever, aktivitet, vad den ska göra vid ett tillfälle
- Många av detta kan vara i olika sjukdomar och standardläkemedel t.ex. kortison
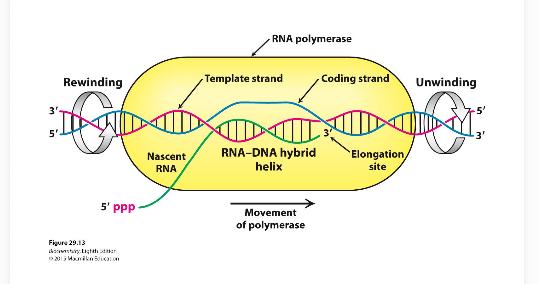 Röd och blå sträng basparar
Gula är RNA polymeras
Poäng är att visa funktion
Skapar nytt RNA (grön sträng)
Utnyttjar basparinformation och gör en kopia i form av RNA
Riktning viktning, dess rörelse, åt höger
Måste sära lite på de här strängarna
Viktig funktion är att separera strängarna så de kommer isär så man kan läsa av och skapa RNA som blir längre och längre allteftersom den rör sig i den riktningen
Sker ei 5→3 riktning
- första nukleotiden som sätts in är i 5’ (röd ppp)
- i 3’ kommer det in nya hela tiden
- för varje protein bildar en fosfordiesterbrygga, som innehåller ett fosfat
- från ATP, 2 ATP spjälkas bort som ger energi i den här processen
- Men i 5’ änden finns det 3 fosfat (ppp)
- VIKTIG POÄNG KOMMER TILLBAKA
- Cellen vet att det är ett riktigt/syntetiserat RNA genom att titta på de tre ppp, då vet man att det är en startplats för transkriptionen
- den gröna strängen är en kopia av den blå, men de läser av den röda
- blå kallas kodande
- röd mall strängen, den man läser av
- man skriver alltid ner kodandesträngen, inte mallen
Röd och blå sträng basparar
Gula är RNA polymeras
Poäng är att visa funktion
Skapar nytt RNA (grön sträng)
Utnyttjar basparinformation och gör en kopia i form av RNA
Riktning viktning, dess rörelse, åt höger
Måste sära lite på de här strängarna
Viktig funktion är att separera strängarna så de kommer isär så man kan läsa av och skapa RNA som blir längre och längre allteftersom den rör sig i den riktningen
Sker ei 5→3 riktning
- första nukleotiden som sätts in är i 5’ (röd ppp)
- i 3’ kommer det in nya hela tiden
- för varje protein bildar en fosfordiesterbrygga, som innehåller ett fosfat
- från ATP, 2 ATP spjälkas bort som ger energi i den här processen
- Men i 5’ änden finns det 3 fosfat (ppp)
- VIKTIG POÄNG KOMMER TILLBAKA
- Cellen vet att det är ett riktigt/syntetiserat RNA genom att titta på de tre ppp, då vet man att det är en startplats för transkriptionen
- den gröna strängen är en kopia av den blå, men de läser av den röda
- blå kallas kodande
- röd mall strängen, den man läser av
- man skriver alltid ner kodandesträngen, inte mallen
- rewinding går igen bakom (början/slut)
 går att se bubblan till höger i blåsträng, ungefär 17 baspar lång
bubblan är temporär, som finns inne polymeraset, så går den igenom bakom och öppnas upp framför
2006 fick någon Nobelpriset för att förklara det här
går att se bubblan till höger i blåsträng, ungefär 17 baspar lång
bubblan är temporär, som finns inne polymeraset, så går den igenom bakom och öppnas upp framför
2006 fick någon Nobelpriset för att förklara det här
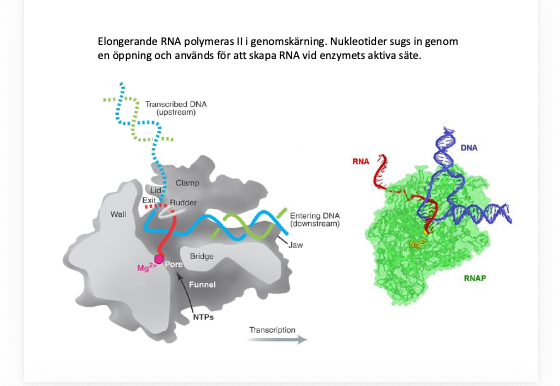
Två viktiga förmågor i RNA-polymeras
- öppna för att komma åt informationen
- hjälper till att skapa den nya kedjan
- har en liten pool, en öppning som suger in nukleotider som en dammsugare, så de kommer in till det aktiva sätet i enzymet
- enzymen kan vara väldigt stora, men aktiva sätet kan vara väldigt litet
- åker in genom en tunnel, precis vid 3’-änden där den nya nukleotiden ska sättas
- kan känna av bassekvenens i den där mallsträngen
- efter reaktionen bildas en fosfyresterbindning
- när trifosfatet spjälkar
- finns det lite joner, Mg2+, kan ibland vara mangan, en liten jon som hjälper till att nukleotiden landar i rätt position

Finns 3 stycken RNA polymeraser
- I + III
- De har en mer specialiserade funktion, bara vissa
- I
- Upphovt till ribosomalt RNA, de som bygger upp själva ribosomen. Den läser av ett antal av de ribosoma RNA
- FRÅGA: Numrerna är hur de rör sig i vissa röra, namnen beror på hur snabbt de sendimenterade, från 40-talet, idag skulle vi aldrig ge de namnen, men de har stannat kvar
- Hände innan man fick reda på DNA koden fungerade
- II
- Pratar mycket om den här för att det är det enzymet som tar hand om skapandet av mRNA
- VI har väldigt många proteinkodadnde mRna som ger cellens olika funktioner
- spelar stor roll fysiologisk
- snRNA kommer vi tillbaka till
- III
- Den läser av ett av det ribosoma RNA, tar också hand om de små tRNA klöver liknande strukturerna, Finns också ett i mitokondrier men det ska vi tala om i T3
Ser olika efekter på 𝛼-amanitin det behöver vi inte komma ihåg
- men vi tar upp det eftersom det slår mot T2, kanske vi stöter på när vi är färdiga läkare, finns i den lömska flugsvampen
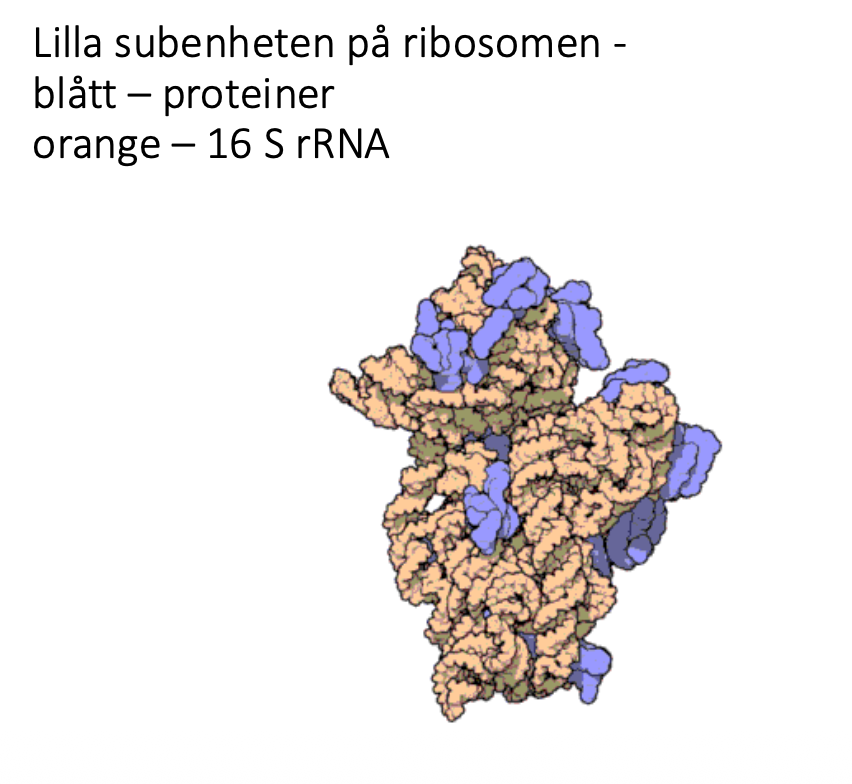 Ute i cytoplasman, det oranga är ribosomalt RNA molekyl, de bildar en gemensam struktur, det är mycket en blandning av proteiner och RNA. När man hör om proteiner och socker och RNA och allt var det är, utövar vissa saker, RNA är en informationsbärare och så. I våra celler används det som finns (det d en har), RNA kan mycket väl fungera som enzymer, finns inga klara/exakta funktioner, grov förenkling att säga att RNA bara är en informationsbärare, den är med i olika processer som man ser ovan
Ute i cytoplasman, det oranga är ribosomalt RNA molekyl, de bildar en gemensam struktur, det är mycket en blandning av proteiner och RNA. När man hör om proteiner och socker och RNA och allt var det är, utövar vissa saker, RNA är en informationsbärare och så. I våra celler används det som finns (det d en har), RNA kan mycket väl fungera som enzymer, finns inga klara/exakta funktioner, grov förenkling att säga att RNA bara är en informationsbärare, den är med i olika processer som man ser ovan
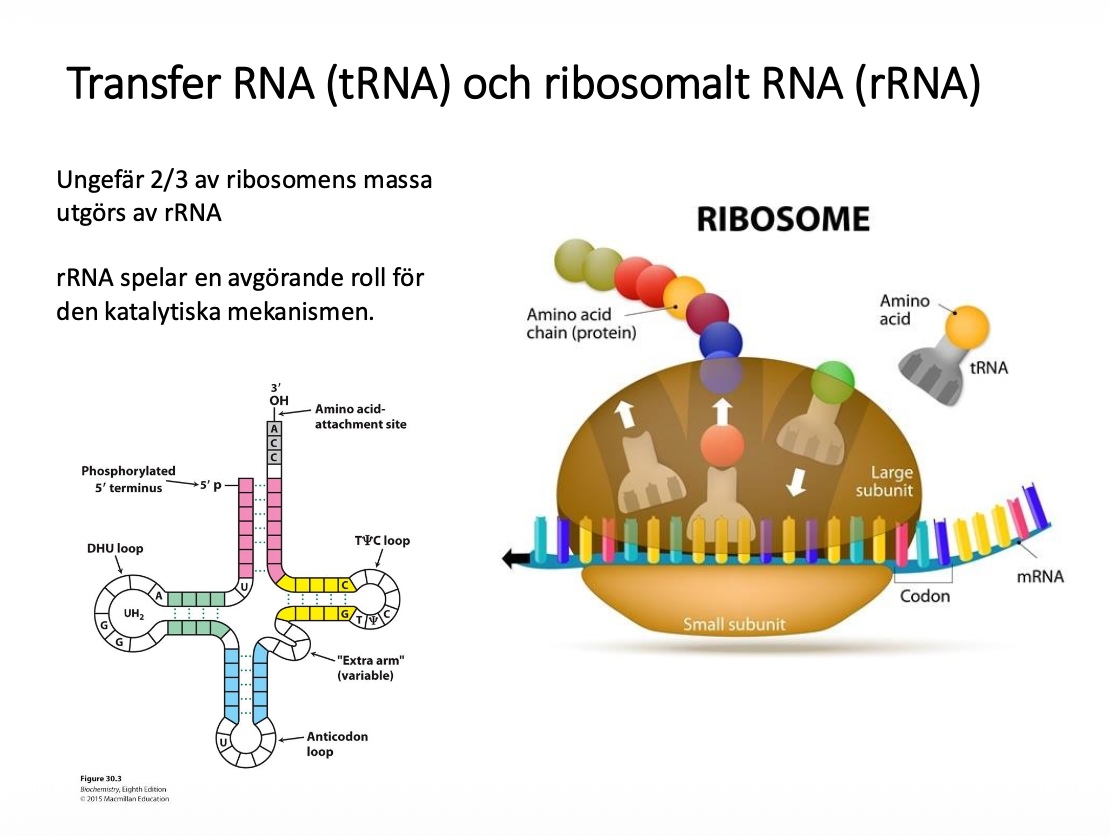 Får mer föreläsningar om detta
Den stora subenheten består av mycket RNA som bygger denna strukturer
Läses av tRNA molekyler, som kommer in och läser av inne i ribosomen.
Får mer föreläsningar om detta
Den stora subenheten består av mycket RNA som bygger denna strukturer
Läses av tRNA molekyler, som kommer in och läser av inne i ribosomen.
RNA är inte bara mRNA, det är också de molekyler som läser av, som också är med och bygger upp ribosomen
 Allt nedanför är polymeras II
Allt nedanför är polymeras II
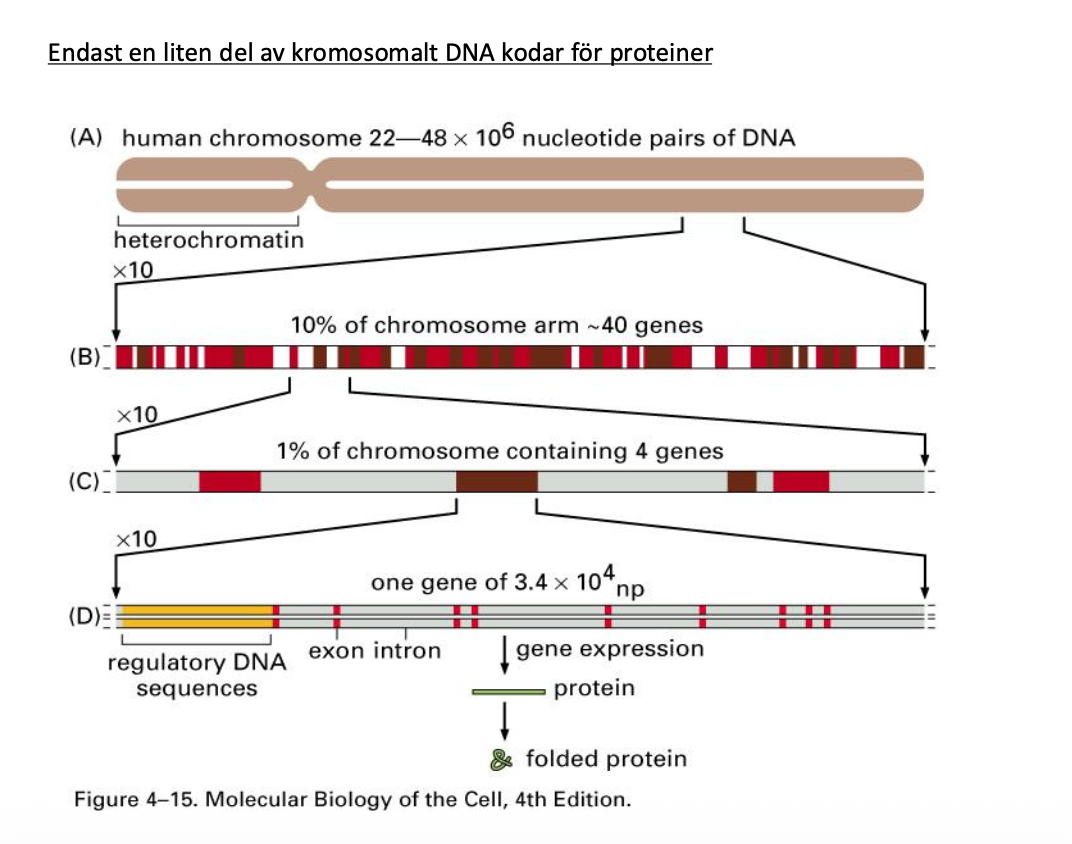
Våra kromosomer är väldigt stora, 3 miljarder baspar är storleken på genomet. Det är en utmaning för RNA-polymeraset att hitta rätt
När man tittar lite mer noggrant, ungefär 98,5% utav DNA de ger inte upphov till några genprodukter. Bara ett icke-kodande historier som finns där. Gäller att hitta rätt i hela det här genomet så man kan starta på rätt ställe. Generena är lite luriga eftersom de finns sällan i ett enda stycke, de kommer ofta i små delar som måste klistras ihop (splicing). Det är en utmanining att hitta rätt.
Kanske börja transkriberas vid orange, kan bara hitta det via de svaga, TATA-boxen, men så svårt att man behöver en enhancer för att hitta det.
 DNA polymeras har svårt att hitta rätt själv, behöver hjälp
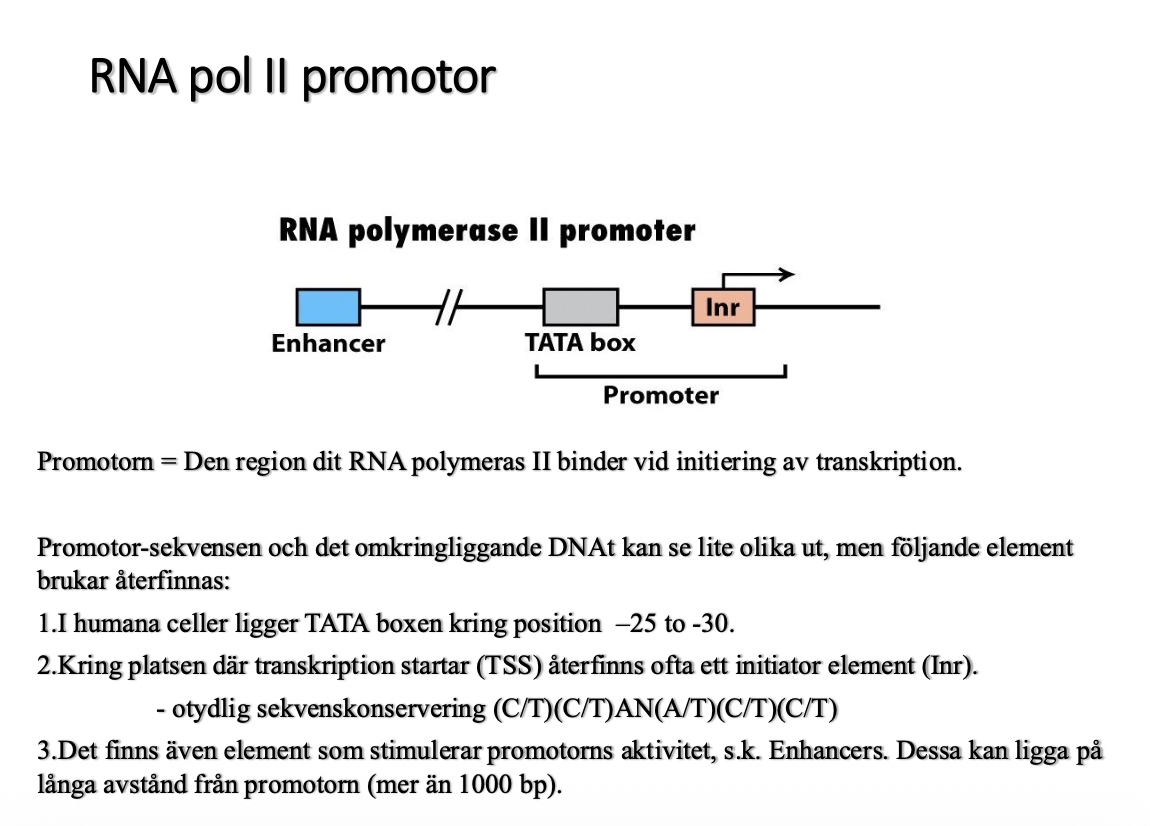
Behöver hitta en promotr
promotor
DNA polymeras har svårt att hitta rätt själv, behöver hjälp
Behöver hitta en promotr
promotor
- är den regionen där poymerasen kan initiera transkription
- exakt hur den ser ut kan variera
- men på sliden finns det den typiska, hur den ser ut
- binder där det står promotor ovanför, för att starta transkriptionen
- man brukar rita det som en pil där det startar, se Inr
- pilen anger var man lägger ner den första nukleotiden
- det är +1, det är det första som läses av och blir RNA
- Åt höger ökar det +10, +20 osv fortsätter, det är ett sätt att veta hur lång RNA-polymeraset har kommit
- Hjälper åt att styra, gasar och bromsar
- Åt vänster går det nedåt, -10, -20. Finns dock ingen nolla!
- varje siffra representerar ett baspar
- Promotorn brukar man hitta vid en TATA-box
- brukar ligga ungefär vid -25 till -30
- TATA-box
- är egentligen bara en bit där det finns mycket ATATATTATA
- AT basbaren är lätta att smälta, då det bara finns 2 vätebindningar
- Inr-element
- baspar där det håller på att start (BEHÖVER INTE KUNNA)
- skiljer sig ganska mycket åt, svårt att se ett speciellt mönster
- inte speciellt konserverad, ser olika ut på olika gener
- Enhancer
- Pratar mer i T3
- Finns stimulator som slår på genbygget, förstärkare på svenska
- Kan ligga mer än 1000 bp ifrån, kan hjälpa till att starta
RNA polymeras II behöver hjälp av följande faktorer för att igenkänna promotorn och initiera transkription • 6 basala faktorer • TFIIA • TFIIB • TFIID • TATA-binding protein (TBP) • TBP associated factors (TAFs) • TFIIE • TFIIF • TFIIH • Uppbyggt av 10 subenheter, bl.a. ett proteinkinas och ett DNA helikas. Kinaser är enzymer som kan överföra en fosfatgrupp (t.ex. från ATP) till proteiner eller andra molekyler.
Essentiella: de behöver man på alla gener, de är essentiella De har lätt, de har hört TranskriptionsFaktor II A, B, D, E, F, H
- C och G har tagits bort, misstag av forskningen
D och H är de viktigaste D:
- första faktorn, det är den som startar
- innehåller flera olika subenheter, RNA-polymeras är faktiskt 12 olika proteiner som bygger upp, stora proteinkomplex
- D är ett exempel består av 13-14 olika
- Proteinet som är intressantat för oss är
- TATA-bindande proteinet TBP
- det är det som binder till tataboxen
- känner igen och binder dit
- TBP associarade kallar man de andra TAFs
- TATA-bindande proteinet TBP
H:
- sista faktorn, som gör att transkriptionen sticker iväg
- Också ett multiproteinkomplex med 10 subenheter
- proteinkinas
- enzymer som överföra en fosfatgrupp på ett protein, eller på ett socker, så det sätter på ett annat protein
- DNA helikas
- det ska vi prata mer om imorgon
- det kan sära på DNA
- behövs för att skapa transkriptionsbubblan, hjälpa till med det dubbelsträngade DNA, så man kan få en bubla
- proteinkinas
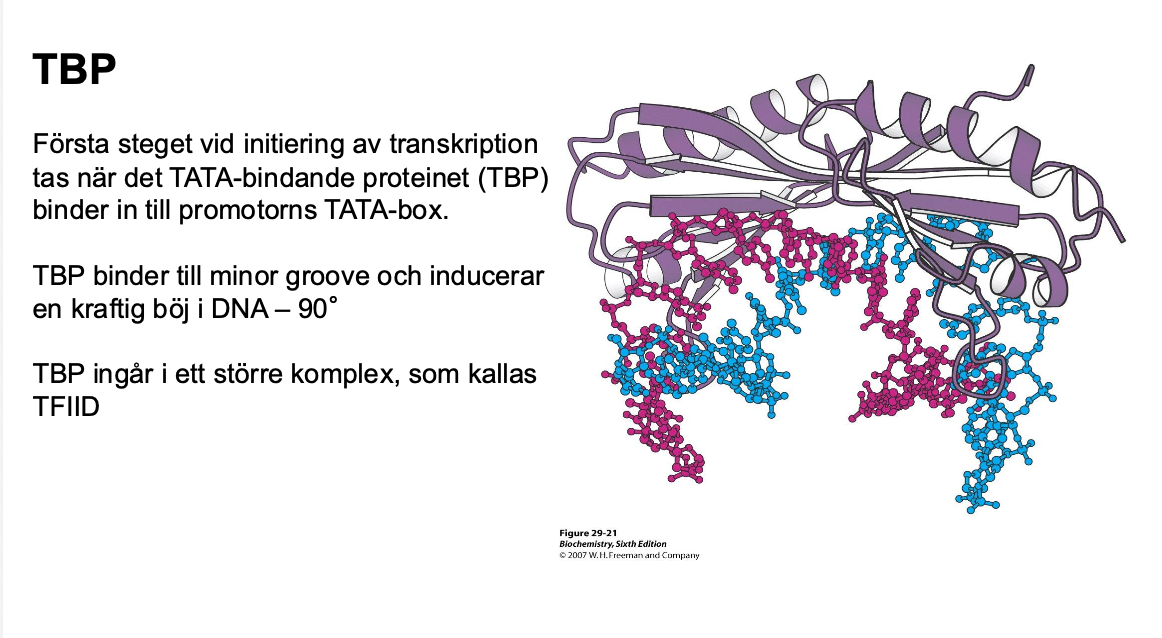 TBP känner igen TATA-boxen, känner igen en viss proteinsekvens, den är lite lustig eftersom den binder till minor groove, det brukar inte proteiner göra, och sen gör den en kraftig böj i DNA, se bilden ovanför, det är i princip 90 grader, sätter sig som en sadel i minor
TBP känner igen TATA-boxen, känner igen en viss proteinsekvens, den är lite lustig eftersom den binder till minor groove, det brukar inte proteiner göra, och sen gör den en kraftig böj i DNA, se bilden ovanför, det är i princip 90 grader, sätter sig som en sadel i minor
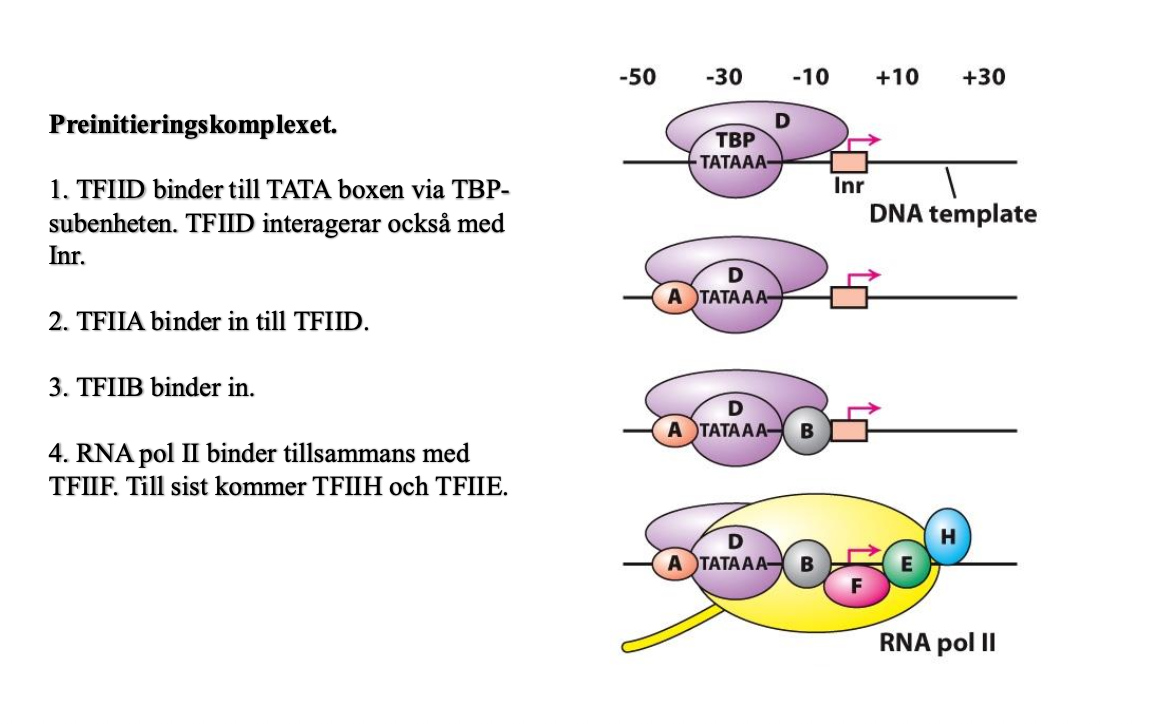 Allt det här sker i en sekvens
Allt det här sker i en sekvens
- det första som händer är att det TATA-bindande proteinet binder till TATA-boxen
- Sen kommer A, sen B osv
- Den sista är H, den kör igång med sin helixas och kinasaktivtet Brukar kallas ett preinitieringskomplex, redo att starta men har inte startat.
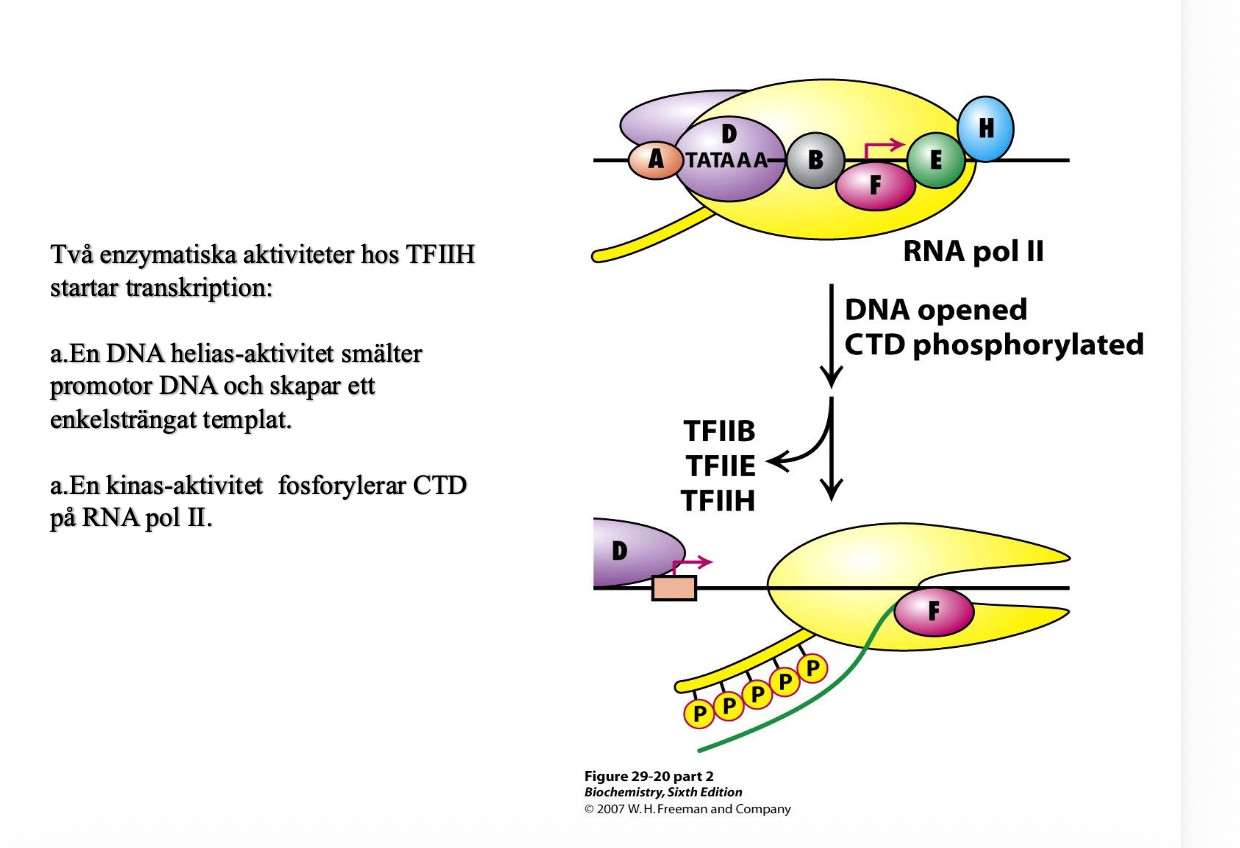
- helias som smälter och skapar enkelsträngat
- kinas aktivtet fosfylerar RNA polymeras II
- så själva RNA polymeras II fosfoluysear så att de släpper och sticker iväg
- man ger gas, färdig och ladda, men för att sticka iväg krävs ett enzym
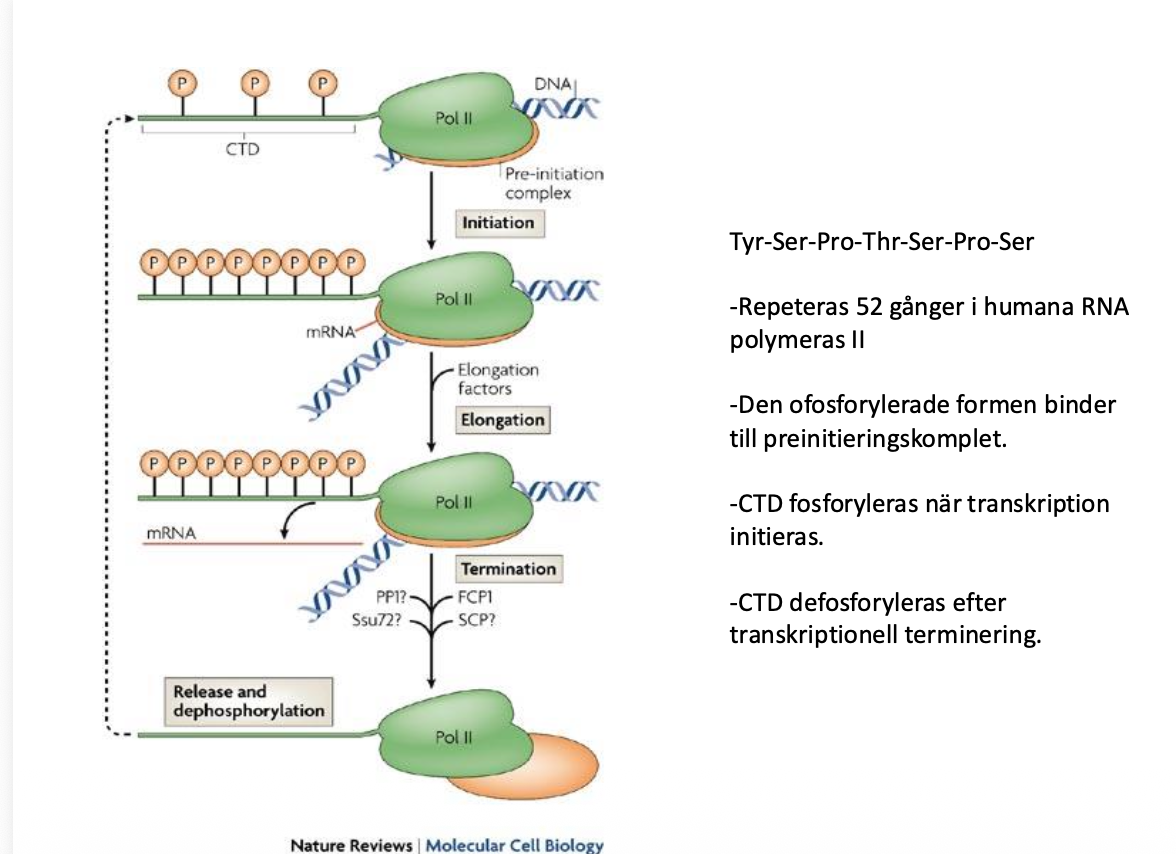
I vanlig fall är det inte mycket fosfat, men sen sätts det på och sen när det går igång stå trillar fosfaten av
fosfaterna sätts inte på var som helst, de sätts på en liten struktur som ser ut som en svans, en lång proteinstruktur och åker efter RNA-polymeraset som en svans, den är faktiskt jättelång det är den svansen som har en ganska otydlig struktur, det är den som repeteras och fosfyleras, beroende på om RNA-polymerases transkriberas eller ej, sätts på av beroende vad som sker. det är en cykel
CTD
- c-terminala domänen är där fosforsvanen sitter
- bildar ingen alpha helix/beta etc, den bara sitter fast hänger efter
- den har massa aminosyror som förstör, den ligger och flackar
- har mycket aminosyror som kan fosfolyserar, massa gånger
- 250 platser som man kan fosfolysera den här svansen, en riktig omfattande
- först är de väldigt lite fosfater, då sticker polymeraset iväg
- när genen är klar trillar de av
- sen ligger det och väntar och laddas
Filmer
https://www.youtube.com/watch?v=SMtWvDbfHLo
Här i börjar TIID med TPB ser ni böjen på DNAt som är rött DEn är bunden och sitter till promotorn sen kommer de andra faktorerna in sen kommer de in binder till sist har man fått in alla faktorer nu kommer en enhancer (pratar ej hur det går till nu) när allt är igång med initierings aktivatorn sen kämnas alla kvar
sen ser man bubblan i slutet
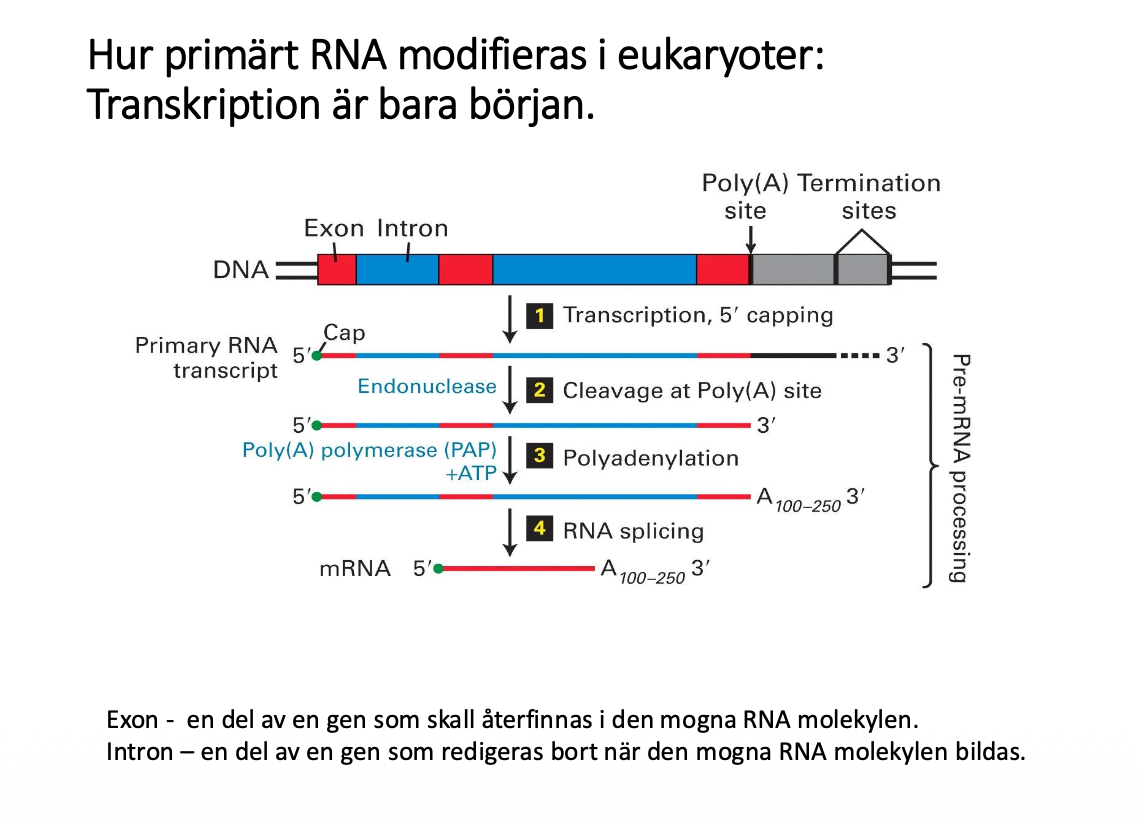
Att avläsa DNA är bara första delen av att skapa ett mRNA De olika stegen när man modiferar eller ändrar brukar man kalla RNA processing gör det färdigt för att det ska vara moget
Sliden ovanför är sammanfattande
Börjar med primärt RNA transcript och läser av hela, startar from promotorn, den här kopian som man ska jobba med
- Sätter på en struktur i 5’-änden en CAP
- Man stoppar transkriptioenn på ett intressant sätt, det är RNA polymeraset i säg stoppar det inte, sen kommer det ut en bit RNA som ser ut på ett rätt sätt, det finns ett enzym som kommer in och klyver det, då känner RNA av att det klippts av och då stannar det
- Sen sätter man på en PolyA-svans som bara sätts på, det kommer inte ifrån genen, man tar bort stora stycken av genen
- Splicing: Innehåller både exon och introner (icke kodande). man tar bort intronen som inte ska vara med
- splitsning när man jobbar med rep, handlar om att sätta ihop de röda exonerna
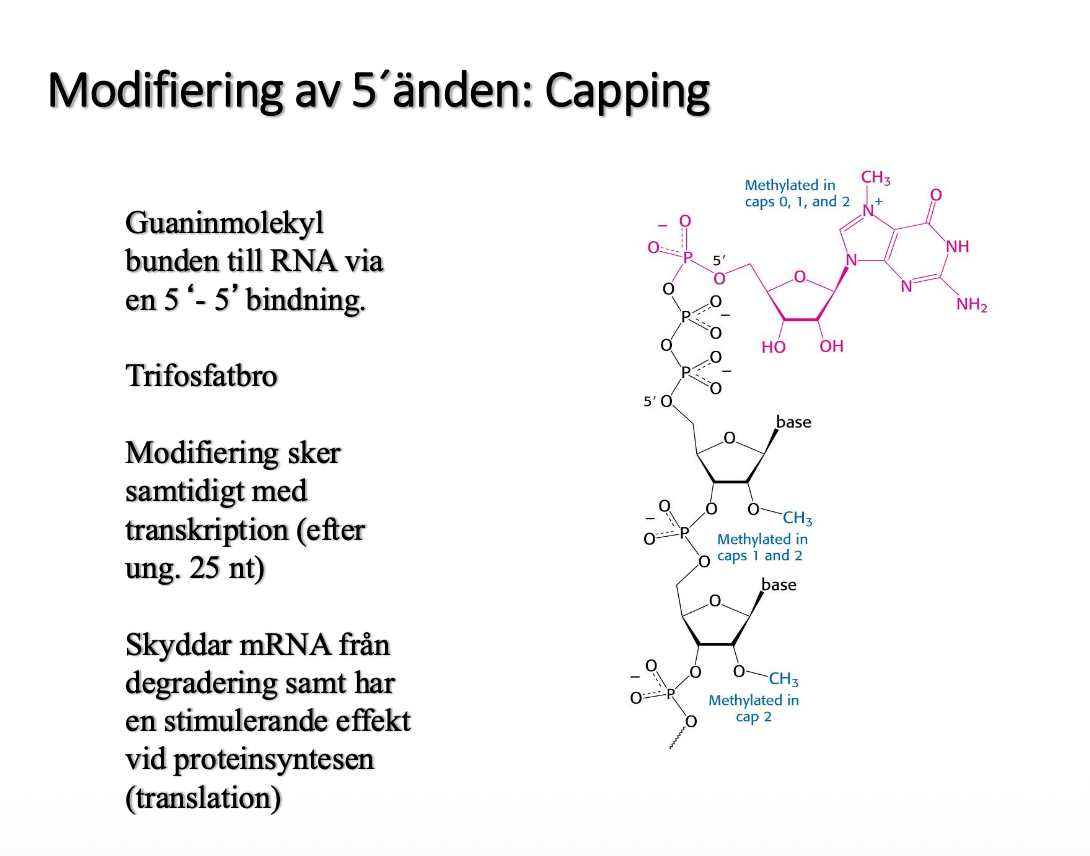
5’ änden har 3 fosfatgruppar, capping enzymer känner igen 5’-änden, det som händer är att enzymerna sätter på en extra nukleotid, en guanin. Den röda uppe i bilden. De känner igen början, det är här vi jobbar, de sätter på den lite lustigt, sätter på den upp-och-ner-på, så är det en 5’-5’ binding Det är en trifosfatbro Det sker väldigt snabbt, efter 25 nukleotider, det jobbar och när det går. Sen direkt sätter den på den lika cappen
Poängen är att
- skydda mRNA så cellen inte gör sönder det, från att degraderas.
- stimulerar även proteinsyntesen
FRÅGA: är 25 hur lång kedjan är inne i polymerasen?
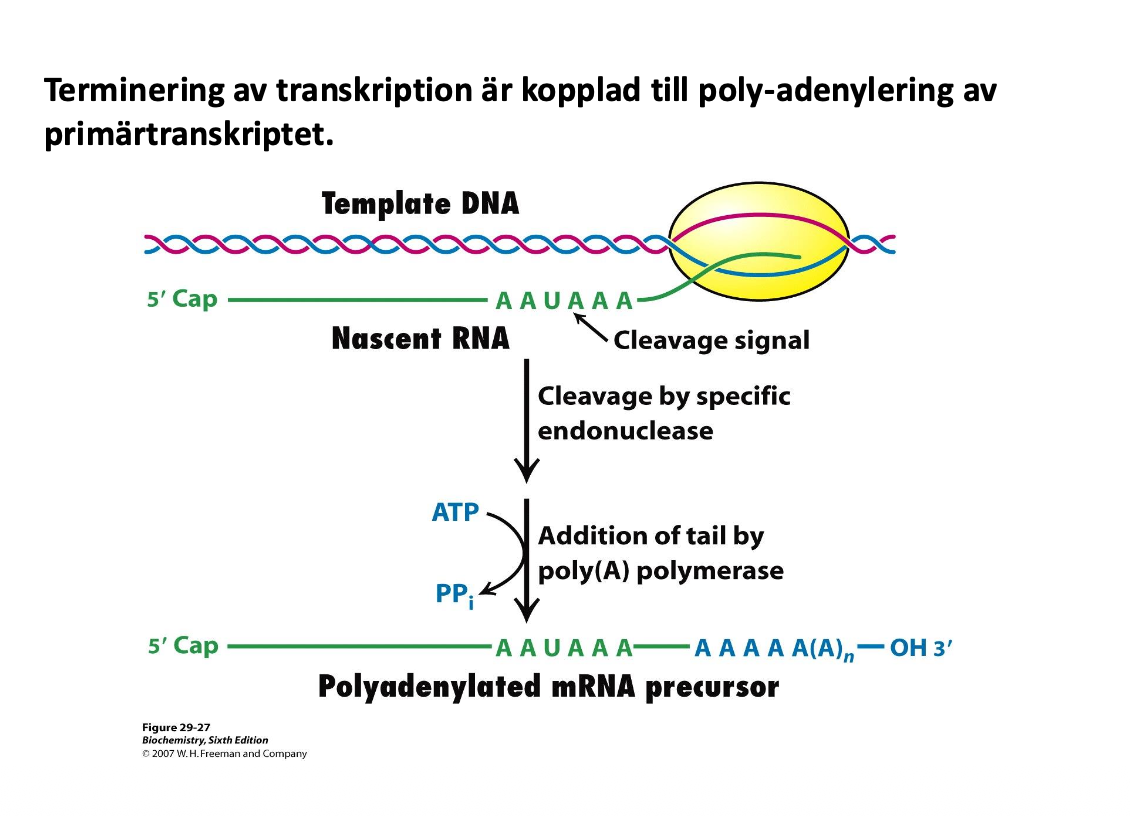 Capping sker först
Sen sker två saker på samma gång och det är att man avslutar transkription /terminerar OCH sätter på en RNA-svans
Capping sker först
Sen sker två saker på samma gång och det är att man avslutar transkription /terminerar OCH sätter på en RNA-svans
RNA kommer ifrån bubblan, det dyker upp en klyvningssignal AAUAAA, då finns det endonukleaser
Introducerar endonukleas/exonukleas och enzymer som utför det
Efter klyvningen frigörs RNA, det stoppar transkriptioen, AAUAAA lockar även tillsig poly(A)polymeraset, som sätter på en kedja AAA() på 3’ änden
FRÅGA: vilka bindingar skapas med 3’ OH gruppen och aminosyrorna?
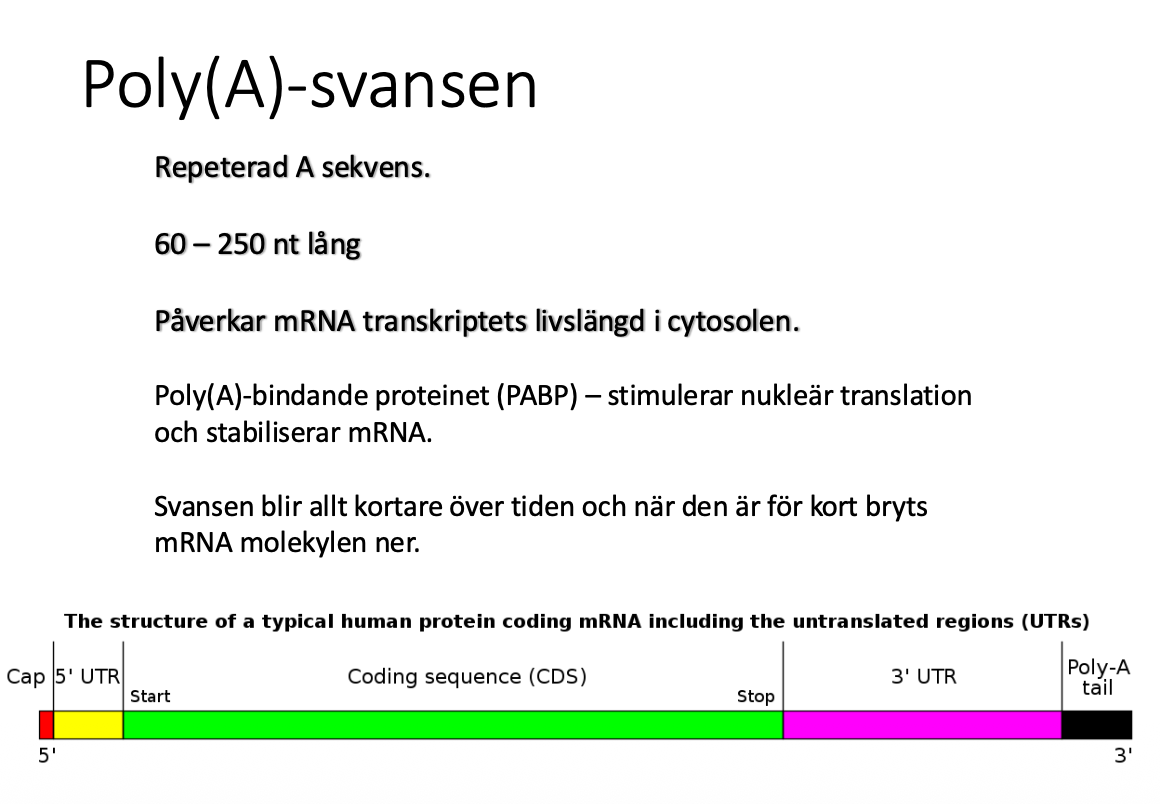 Svansen anger RNAs halveringstid i cytosolen
Svansen anger RNAs halveringstid i cytosolen
- viktigt sätt att reglera hur mycket proteiner man ska få Ju äldre RNA det har funnits, det kortare blir svansen. T3 förklarar mer
Det stimulerar och translation. Både capen och dna-svansen.
Finns alltid en sträcka i början och slutet som inte är med i proteinbilden, de untranslated regions (UTR), finns alltid sådana regioner.
Struktur
- cap
- 5’ UTR
- Coding Sequence
- 3’ UTR
- PolyA-svans
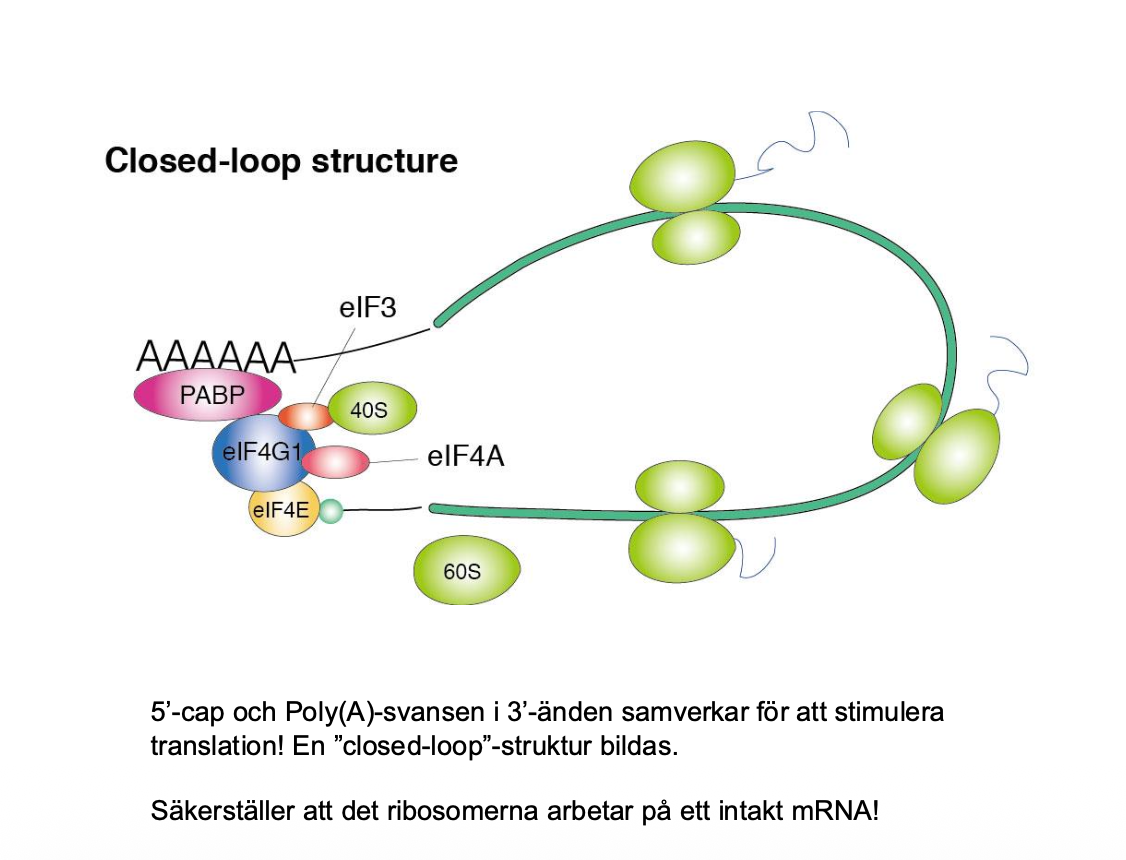
Vi har en cap, den gröna saken. De kommer ganska nära varandra när ribosomen ska börja använda, ribosomen kan veta att det är ett mRNA, men den vet också att den här dna molekylerna har båda delarna. Man kollar att det finns en giltlig cap och en svans (PolyA), måste finnas båda för att det ska vara värt Det heter closed loop structure, säkerställa att ribosomerna arbetar på en intakt sekvens
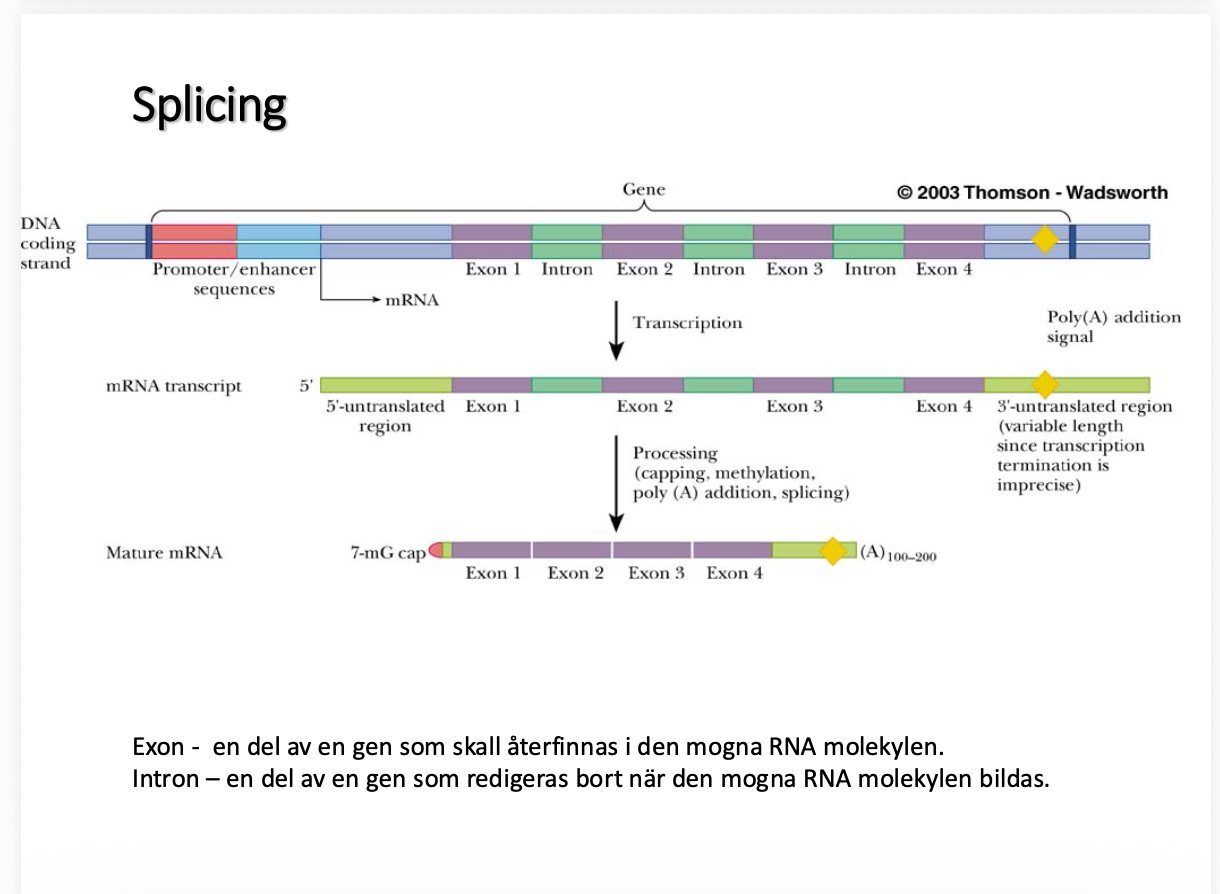
Gener kan vara väldigt lång, de finns icke-kodande delar som eter introner som ska tas bort det som vi ska behålla heter exoner de delar som ska finnas kvar och få en fungerande RNA molekyl
I bilden ovanför ser man hur intronerna försvinner, sen har man cap och svans, det är den mogna
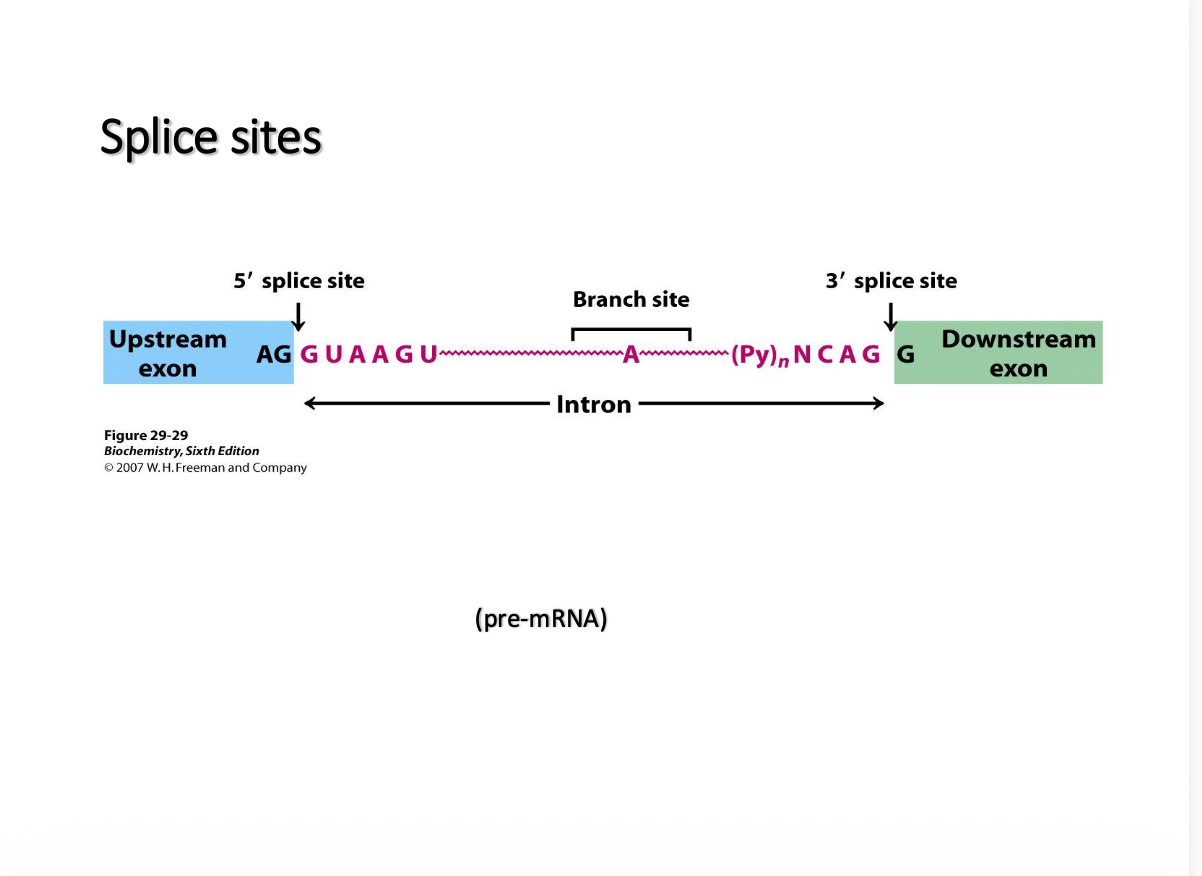
Till skillnad från transkription, eftersm de har konstiga sekvenser. Det har dock inte introner, det finns väldigt specifika sekvensker vad introner börjar och slutar, man burkar kalla det för splice site ska tas bort från 5’ slice site till och med 3’ splice site
Man vet att det alltid finns någonting som är ett greningsställe (branching), det är alltid ett A, det är det som är ett intron, det är lätt att hitta det, t.ex. via datorn. jättelätt att hitta.
viktigt att det sker på rätt sätt, att intronerna tas bort annars kan de ge massa olika sjukdomar.
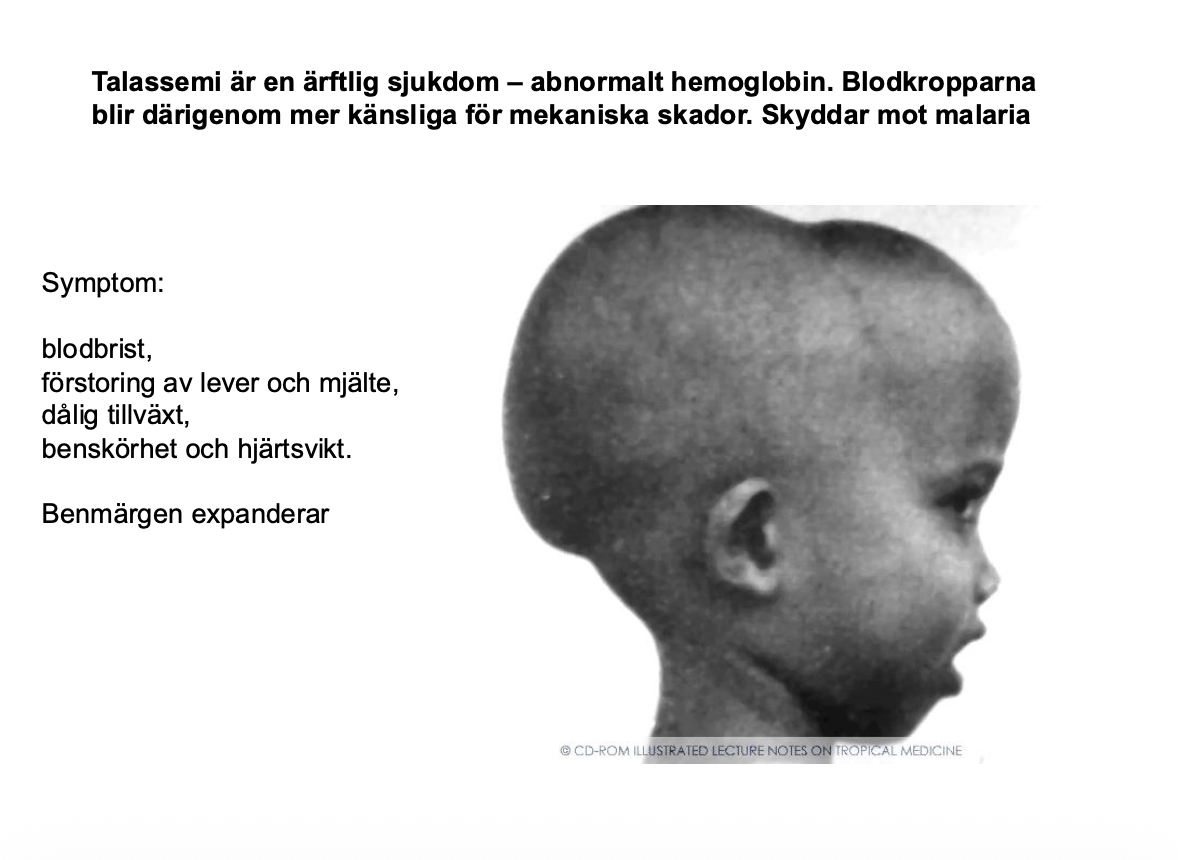 Ett exempel är talassemi är en ärftligt sjukdom, ett abnormal hemoglobin.
Lite som sickle-cell-anemi lite samma sak, att man stör funktion hos blodkropparna, men det är inte bara negativt. Men den råkar skydda mot malaria, då kan det vara en sjukdom kan vara ett jätteproblem för individen men samtidigt skyddar mot malaria
Men de finns oftaas för att de också har en positiv effekt
Ett exempel är talassemi är en ärftligt sjukdom, ett abnormal hemoglobin.
Lite som sickle-cell-anemi lite samma sak, att man stör funktion hos blodkropparna, men det är inte bara negativt. Men den råkar skydda mot malaria, då kan det vara en sjukdom kan vara ett jätteproblem för individen men samtidigt skyddar mot malaria
Men de finns oftaas för att de också har en positiv effekt
blodkropparna fungerar inte som de ska, de ge upphov till blodbrist eftersom de förstörs, kroppen kompsenser genom att bilda mer blod, mer benmärg, då börjar man få benmärg som växer till, det kan växa till lite överallt, i benen då blir man benskör eller ben som inte brukar ha benmärg, t.ex. skallben men det här barnet har talassemi och för att kompensera för de låga blodvärderna så trycker kroppen upp benmärgen och då får man de här förändringarna i skallbenet, det är det som ger benskörhet.
förstoring av lever, mjälte sen får man hjärtsvikt för mer blod behöver pumpas
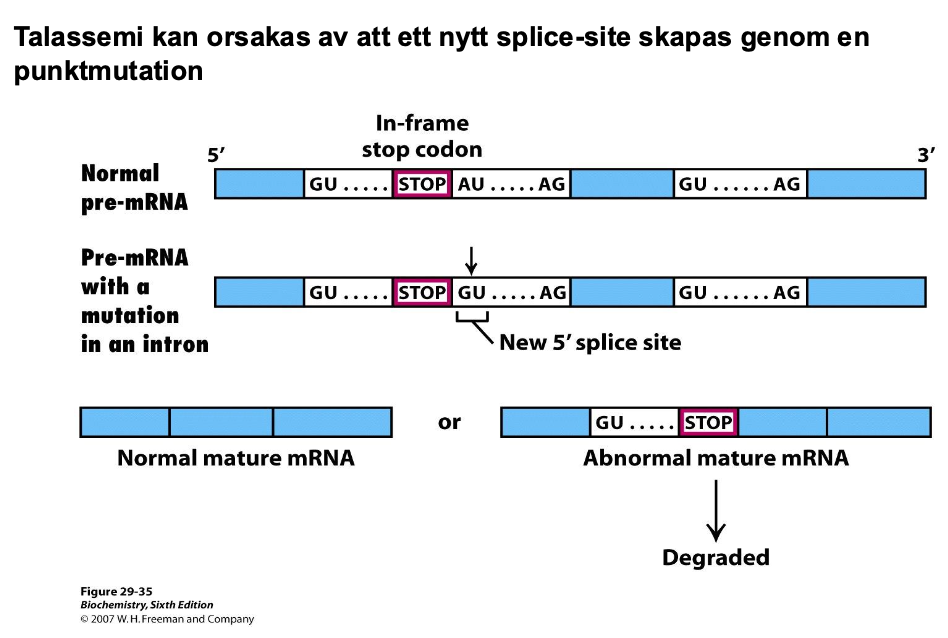
Talassemi orsakas av att nytt splice site, som är felaktig genom en punktmutation.
Man kan inte anväanda intronet som en kodingsekevens, det kan finnas t.ex. ett STOP kodon inne i intronen, man får en mutation inne i introen, så det ser ut som ett nytt splice site, men nu dyker det upp ett nytt här inne och man får en mutation. Kan ta bort intronerna, missuppfatta och får en felaktig splicing som gör att man behåller en del av intronet. Behåller en liten del av det och då får man inte hela proteinet. Men det kanske inte händer hela tiden, kanske bara 70% men det räcker för att få den här sjukdomen
Det är en relativt enkelprocess
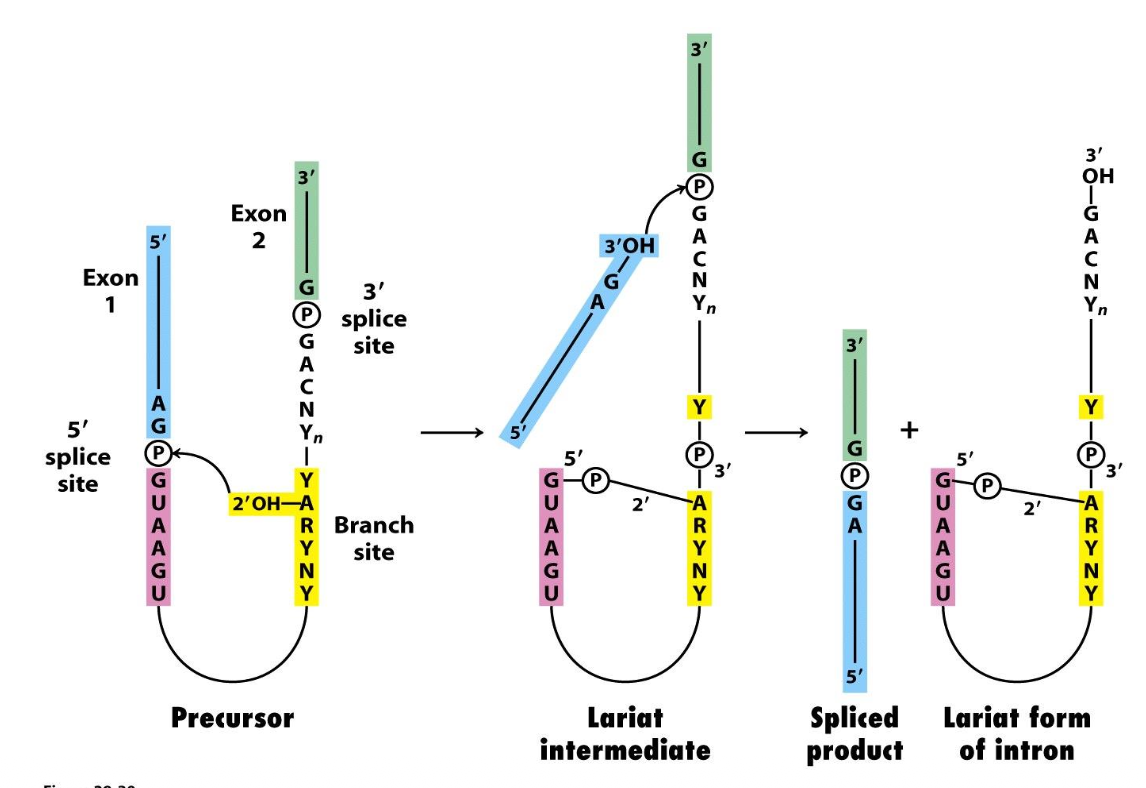
Vi har exon 1 och exon 2 som vill ta bort intronet som ligger imellan, man gör så att A ligger nära greningstillfället. När det böjs på det här stället, så kommer det spontant att ge på den här fosforyesterbryggan, det går att göras på egen hand utan en enzym, det är ovanligt men det finns. i 2’ OH vid A är det inblandat på båda sidor, det är ledigt och kan binda till splice siten då kan den reagera, då bryts bindningen. På det sättet frigörs det här exonet och då har det en fri 3’-ände, då slår det upp och vänder. Det är två fosforysesterbryggor som byter plats, då kopplas exonerna ihop och det som är kvar är bara resten, det är bara en lassostruktur, bara ett larealt. KORT: Splicing handlar om att böja saker så det hamnar rätt
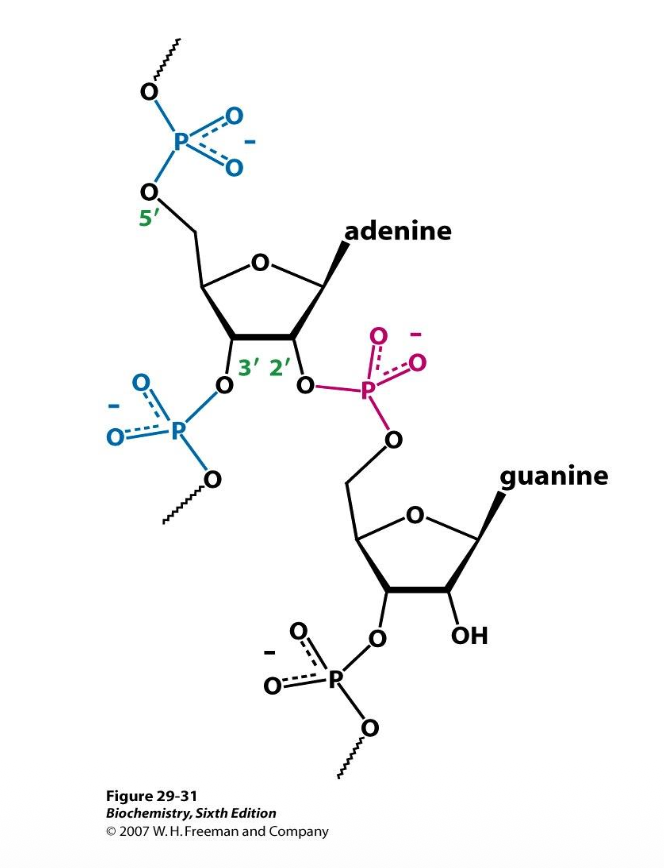
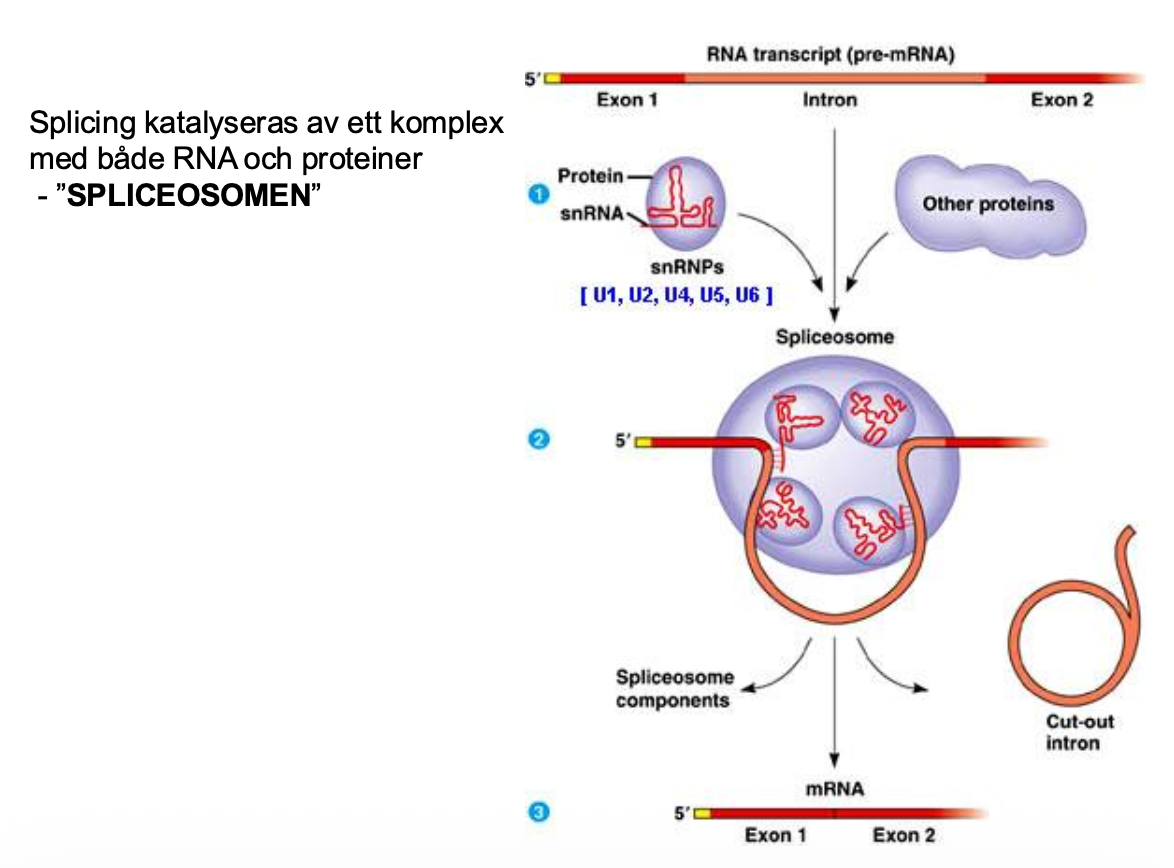
Det finns ett proteinkomplex som heter splicozomer, den hjälper till att böja RNAt så den här reaktionen kan se. Hur hittar den intromerna så exakt? Den har med så små RNA-molekyler själv, består av proteiner och RNA. Det är de som kallas snRNA - small nuclear RNA. De har förmåga att baspara. Den hittar 5’ och 3’ och binder sen sker reaktionen spontant.
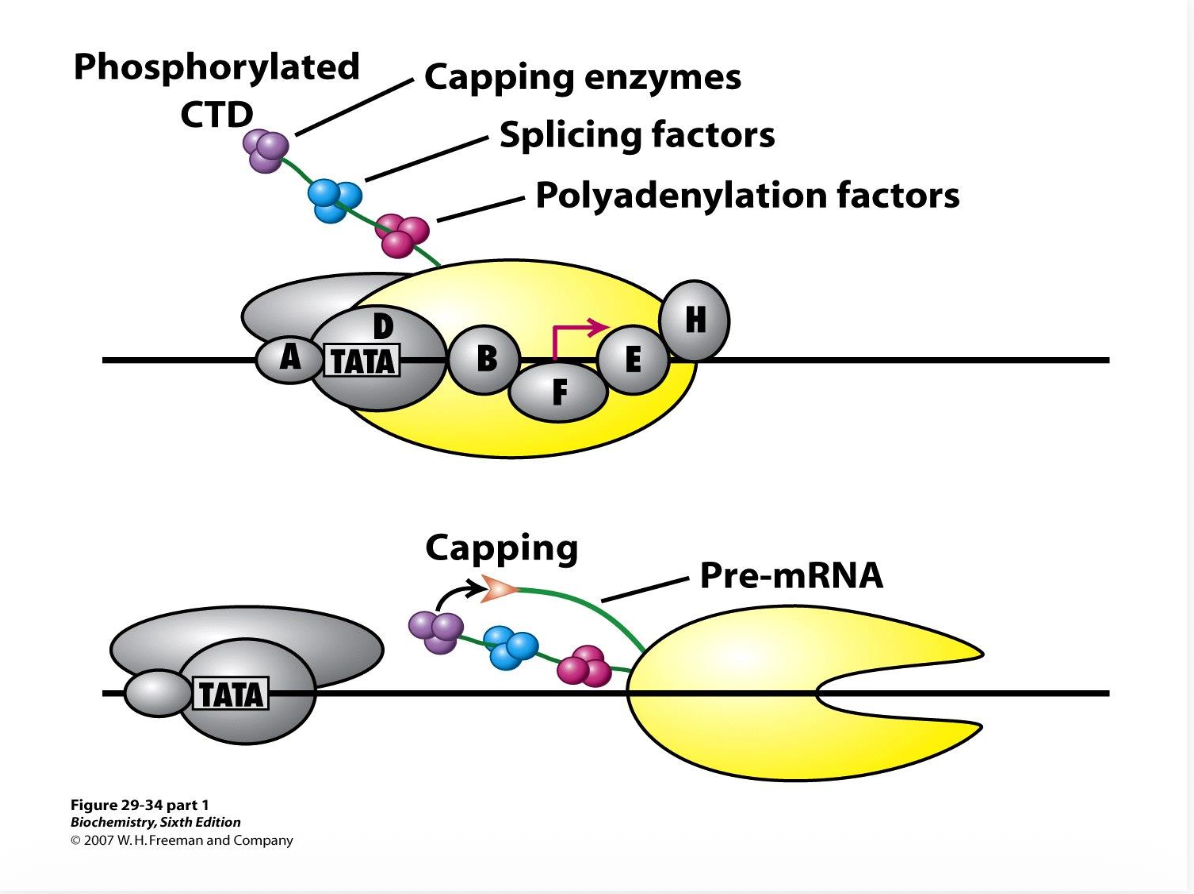
Den långa svansen på C-terminalen spelar en roll, det visar sig vara en platform för capping/splicing/polyadenering kan alla binda dit, om vi tänker på att vi ska göra det här processerna, är det viktigt att faktorerna hittar rätt, ett sätt att hitta rätt är att de binder till RNA-polymeraset, de följer med på ryggen på den långa svansen, då åker det med. De är färdiga och är på plats. när RNA börjar dyka upp så är det väldigt lätt att de dyker upp. De första som händer är att de cappas, sen splicar det
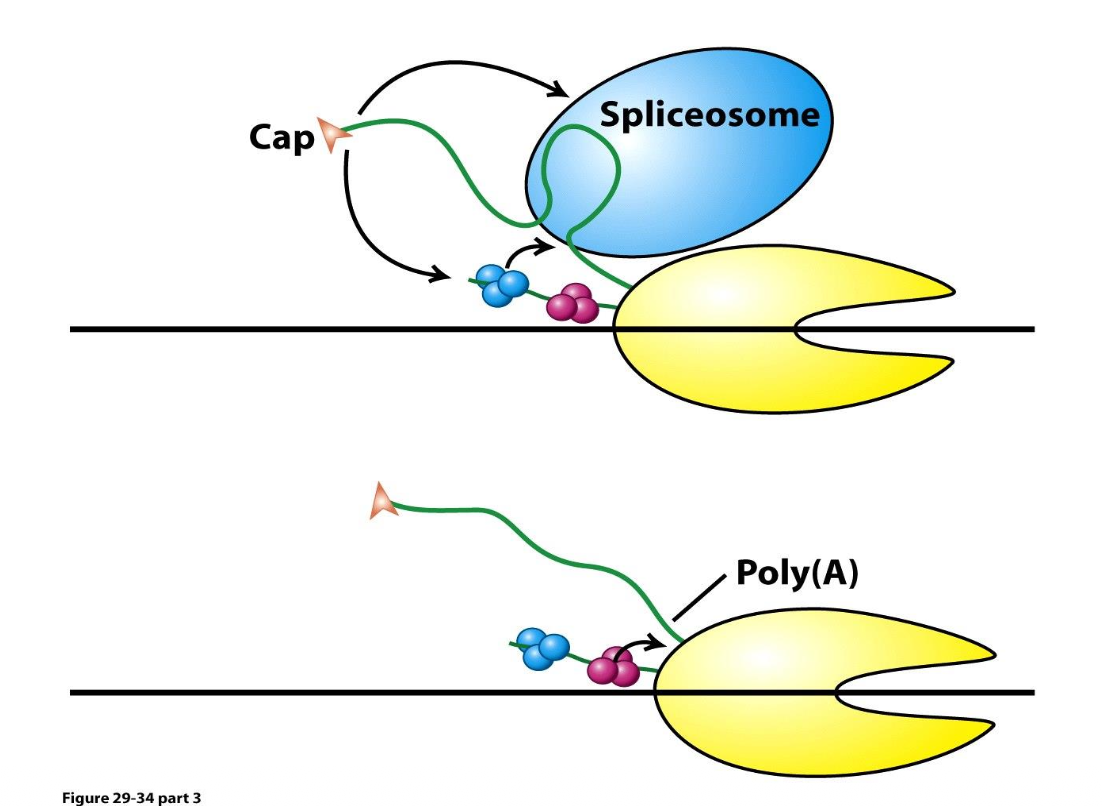
den c-terminal svansen är en plattform för de enzymer som behöver jobba på det omogna, de slipper leta, det är redan på rätt ställe så gör det mycket enklare att utföra det här.
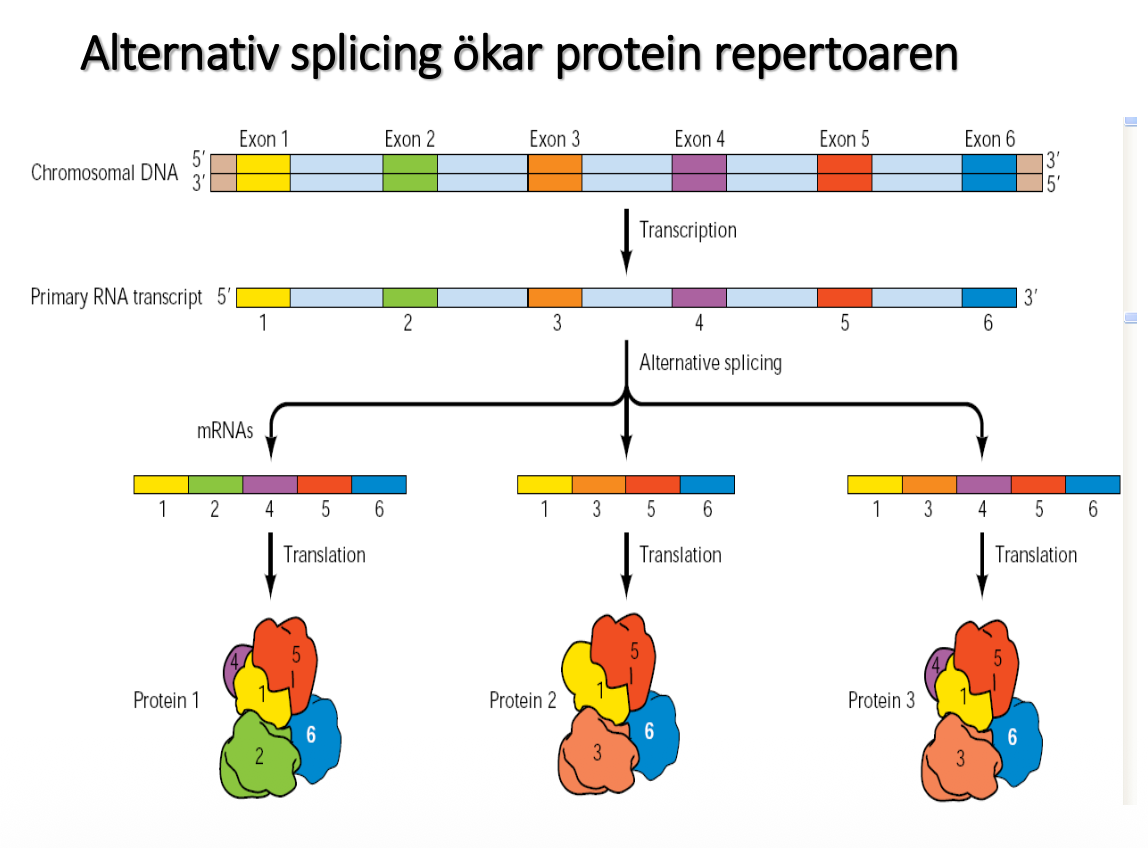
Varför har vi intron överhuvudtaget? Det gör att vi kan väldigt många exon och att vi från en och samma gen skapa olika produkter. Det finns något som heter alternative splicing, det kommer med på T3.
Man behöver inte använda alla exon alltid, man kan välja
- 1-2-4-5-6
- 1-3-5-6
- 1-3-4-5-6
50000 gener, men 200000 gener, olika mRNA
det går aldrig att ändra ordningen, den är alltid samma.

I våra hörselkanaler har vi 500 olika mRNA varianter, ni som inte hör perfekt har problem med splicing. Felsplicade.
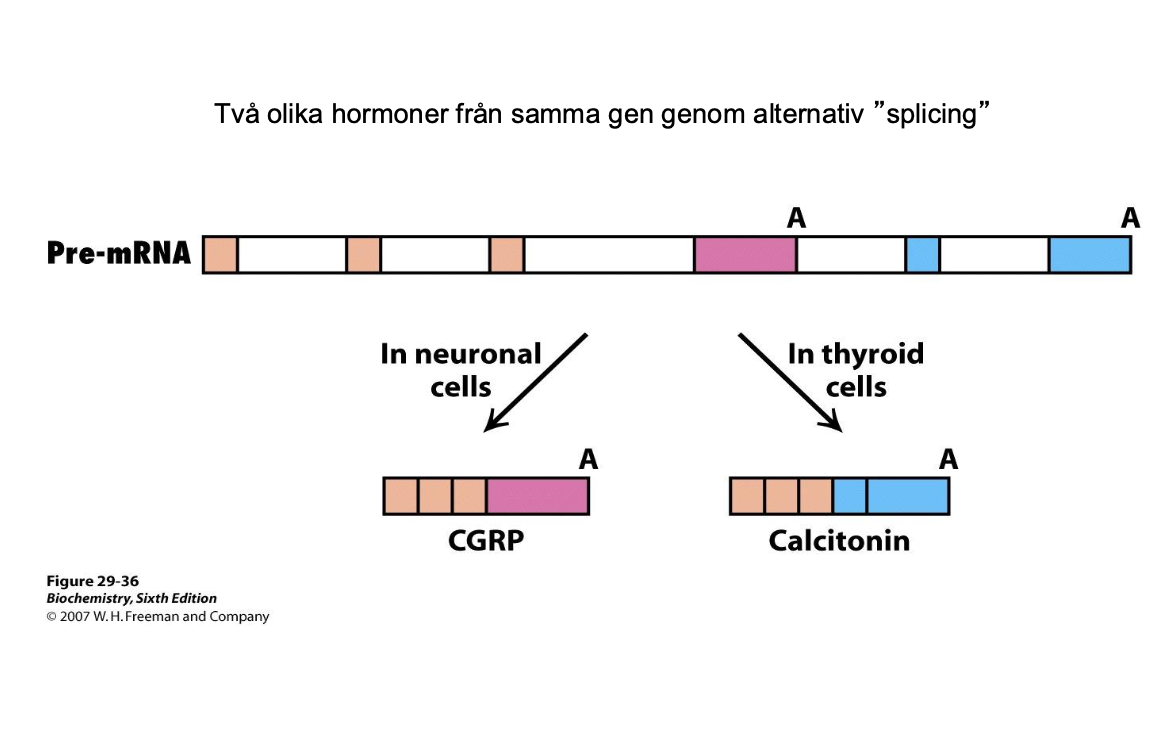 Samma sak för olika vävnader
Samma sak för olika vävnader
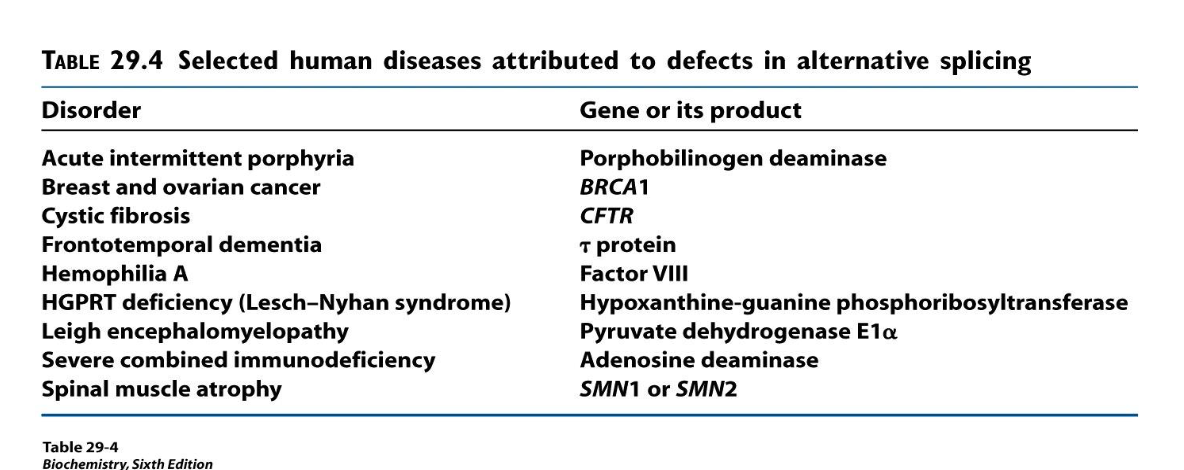
Splicing relaterade sjukdom, som kan ge upphov till allvarliga sjukdomar. Demens, blodsjukdom osv Vissa saker kan man behandla genom att styra splicingen