- En jämförelse mellan eukaryota och prokaryota genom
- mycket är lika!
- Grundläggande enzymatiska processer vid eukaryot DNA replikation (mycket lik prokaryot!)
- dyker upp i läkemedel
Föreläsningen täcker bl.a.:
- Leading och Lagging strand
- Processivity och proofreading
- Replikationsgaffel och replisome
- Superhelicitet och topoisomeraser
- Nukleaser och ligaser
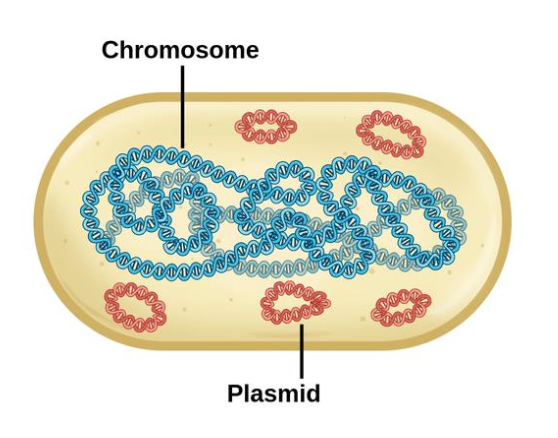 Bakterier har eget genom
Bakterier har eget genom
- Cirkulärt
- vissa problem med linjär replikation av ändar
- fördelar, bara gå runt
- 500kbp till 11Mbp
- Plasmider
- samma typ av DNA, mindre och kan överföras mellan baktirer t.ex. antibiotikaresistens
- Har ett genom
FRÅGA: varför har människor inte plasmider för att överföra fördelar?
Eukaryota
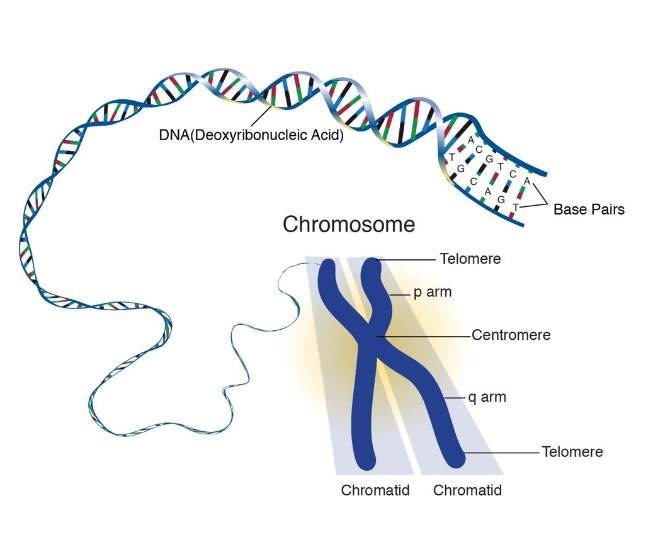
- 30-100 ggr så större än bakteriella
- 3 Gbp
- Egna problem att det är så stort och linjärt
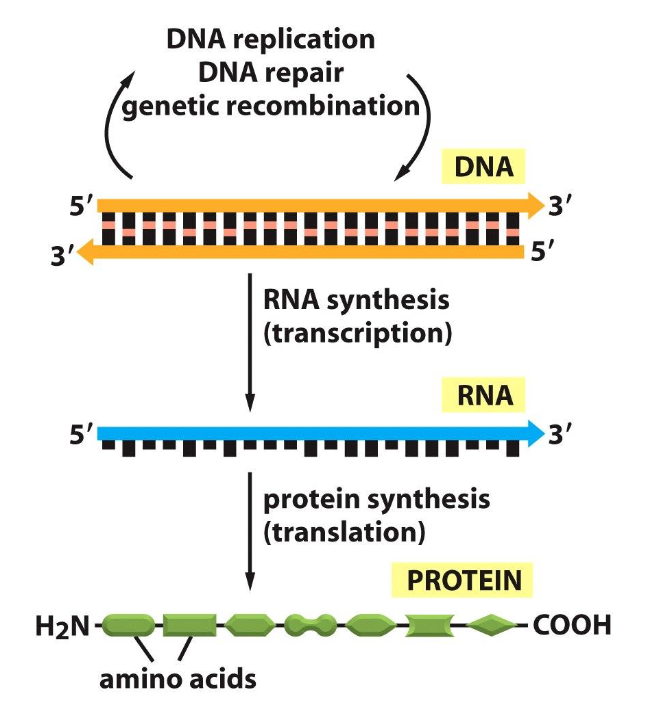
Bra att lära sig:
- DNA replikeras
- RNA transkription
- Protein translation
Redan Watts insåg att det var semi-konservativt så fort de förstod hur det såg ut. Då blev allt väldigt uppenbart
Genom att titta så förstod man funktion, var komplimentära, samma information fanns i båda molekylerna
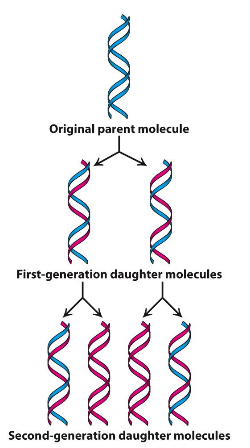
Viktigt:
- semi-konservativ som begrepp
- föräldrasträngen är den blå (parental)
- nybildadsträng är den röda (nascent)
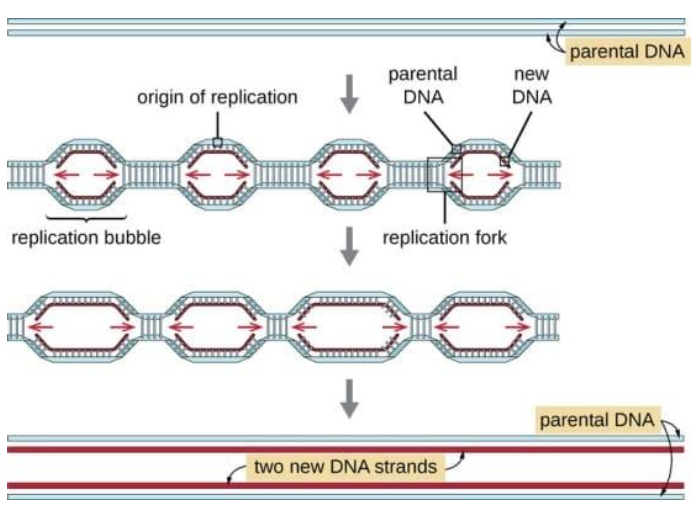
Krångligt att rita ut helixarna, blir linjärt för förenkling
Man börjar på många olika ställen
- bildar replikationsbubblor
- sen växer bubblorna åt båda hållen
- så småningom når det varandra, då går de ihop och då får vi nya strängar
Bubblorna startar vid “origin of replication” och är många, som är väl definierade, väl reglerade Många sjukdomar beror på för mycket/lite dna, eller skapat dna, jättereglerat, en gång, det måste ske samtidigt över hela molekylen.
Varje bubbla har två replikationsgafflar
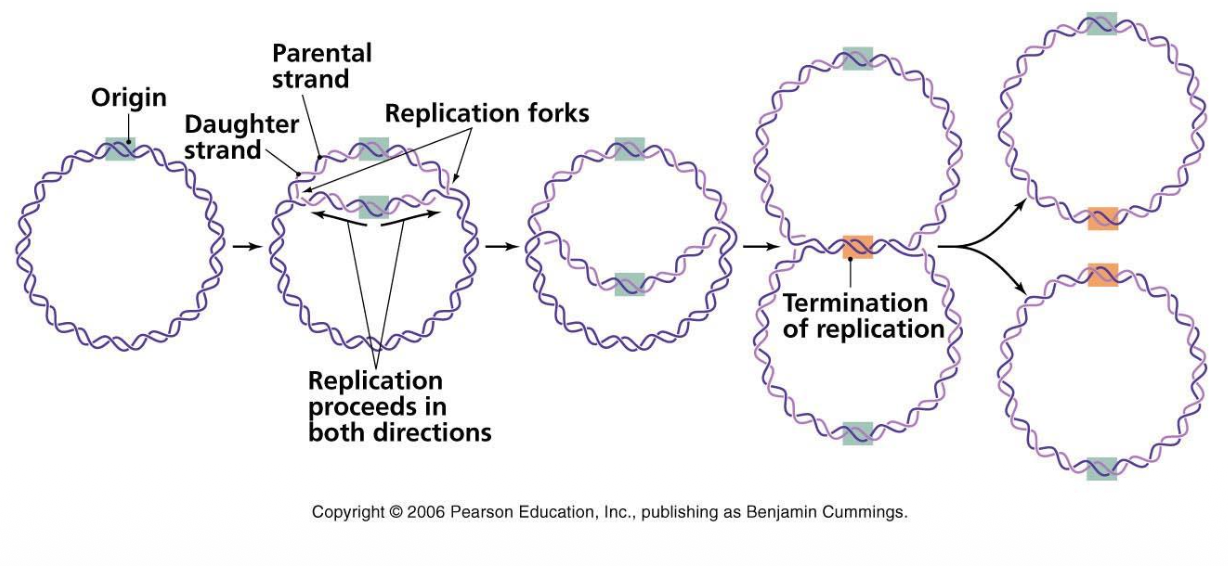 Bakteriell DNA behöver inte så många replikationsgafflar
Har bara ett origin of replikation
Bakteriell DNA behöver inte så många replikationsgafflar
Har bara ett origin of replikation
Hur går det till att läsa av båda två strängarna i samma riktining?
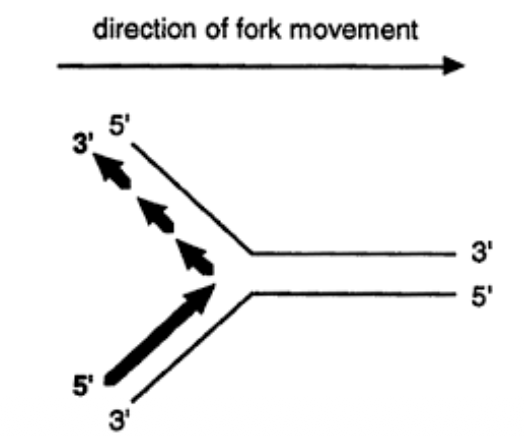
- föräldrasträngarna är antiparallela (övre)
- undre är inga problem, mall och så bygger man en i taget
- Mallsträngen går från 3’ till 5’ felaktig
- behöver en nödläsning
- gör korta snuttar
- lägga en ny som går bakåt
Så
- en kontinuerligt
- en med små korta snuttar
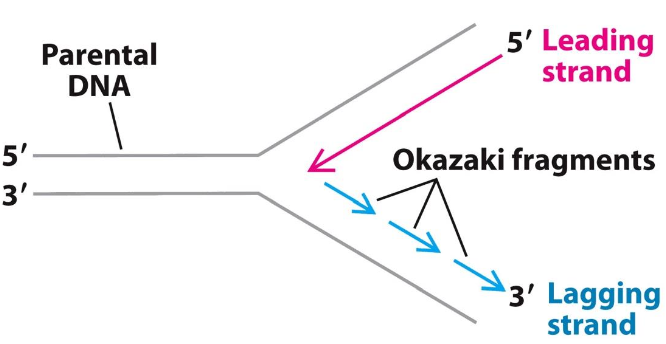 Man brukar prata om två olika
Man brukar prata om två olika
- kontinuerligt: leading strand
- små fragment: lagging strand
- varje fragment heter okazaki fragment
- döpt efter en japansk gästforskare på stanford
- 100-200 nukleotider i eukaryoter
- 1000-2000 i prokaryoter
FRÅGA: varför storleks skillnad mellan eukaryoter och prokaryoter?
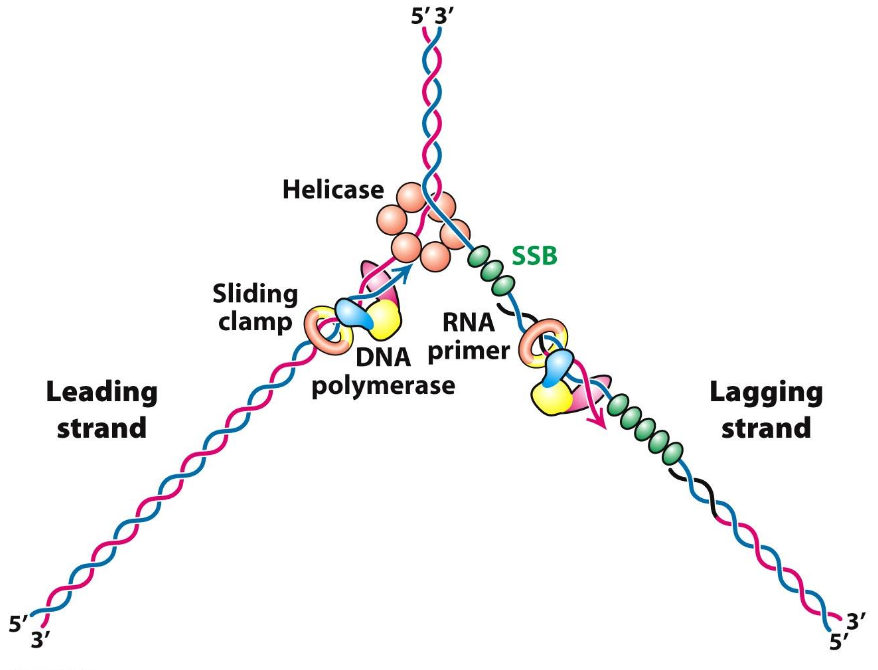 Måste ske av ett helt enzymmaskineri
För att lyckas så måste vi ha ett antal enzymer
te.x ett som hjälper till att dela
skillnad mot RNA:
Måste ske av ett helt enzymmaskineri
För att lyckas så måste vi ha ett antal enzymer
te.x ett som hjälper till att dela
skillnad mot RNA:
- bubblan öppnar bara upp en liten bit
- kräver mycket mer kraft krävs (helikas)
- polymeras för att sätta på
- krävs en för både lagging och leading
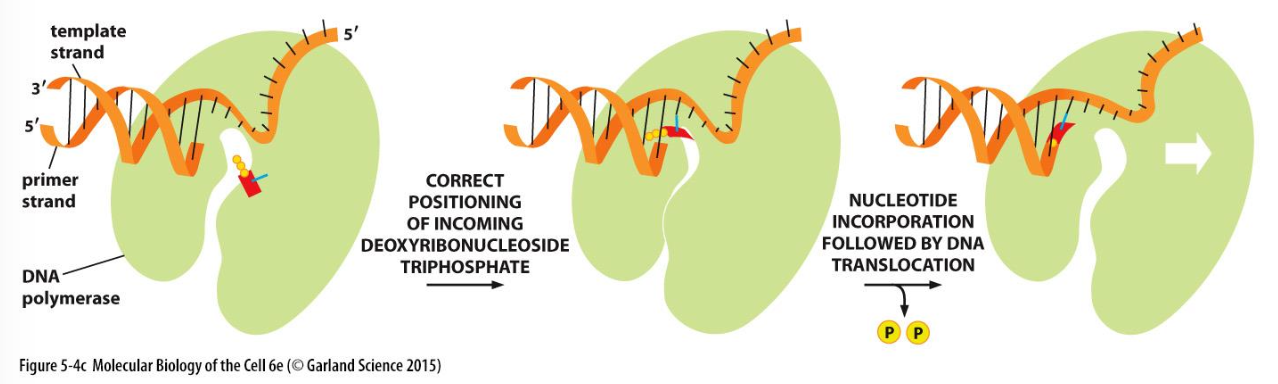 Viktigaste enzymet!
DNA-polymeras
Viktigaste enzymet!
DNA-polymeras
- hjälpa till och skapa en struktur
- nya nukleotider som kan baspara
- kallas DNA-syntes
- DNA-polymeraset hjälper till att välja ut rätt nukleotid
- kan hamna snett, när det inte sitter perfekt så bildas det inget nytt fosfodiesterbindning
- när rätt nukleotid är på plats, nu verkar allt stämma då sker det en liten ändring i strukturen - click säger det och kopplar på i den nya kedjan
- får energi av fosfatgrupperna som trillar av ATP
- då öppnar sig DNA-polymeraset och rör sig en liten bit
FRÅGA: krävs det ett ATP per replikerad nukleotid?
Magnesiumjoner hjälper DNA-polymeraset att sätta fast nya nukleotider SLIDE SAKNAS
interaktion med magnesiumjoner, hjälper till att lägga så allt ligger väldigt nära varandra det hjälper till att få en reaktion skapa en ny fosfodiesterbindnig
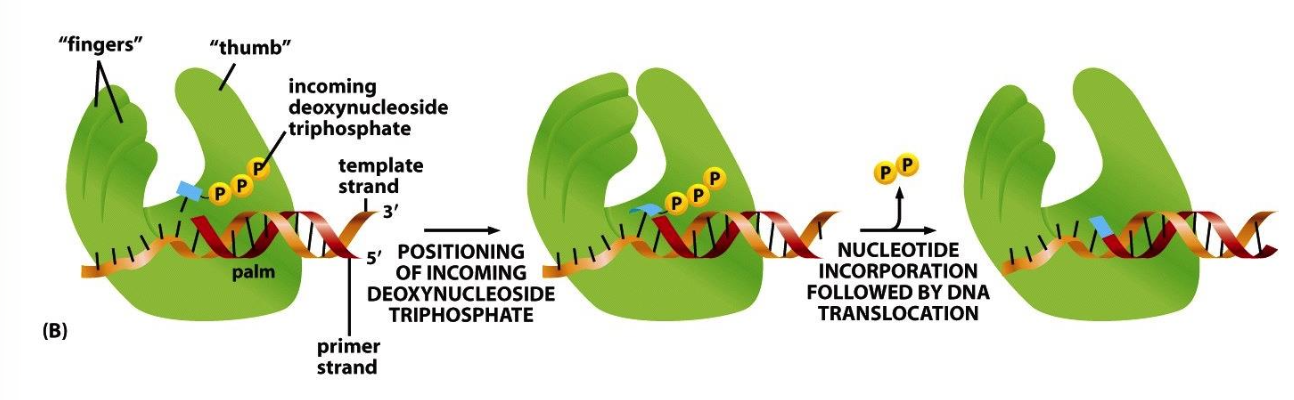 påminner om en högerhand som växer
påminner om en högerhand som växer
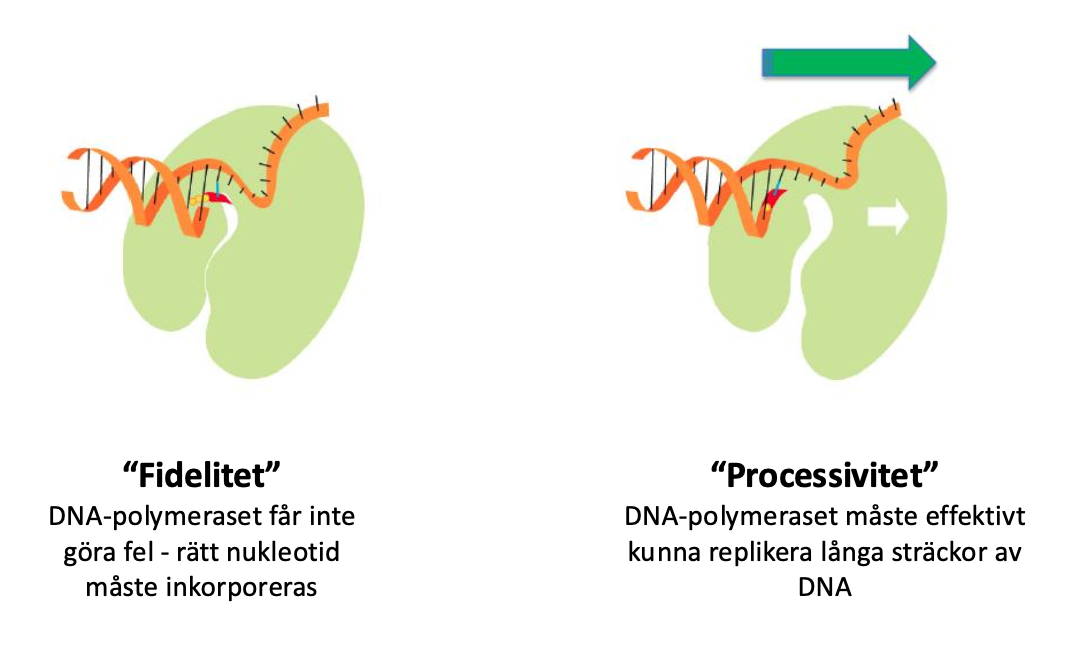 kärnpunkten i vad DNA-polymeras behöver göra
kärnpunkten i vad DNA-polymeras behöver göra
När det inte funkar kan man få väldigt tråkiga sjukdomar, t.ex. sjukdom vid 13 och går bort vid 20. I mitokondrier, talar mer om det i T3
Cancer orsakas pga av mutation, då man inte kan mutera DNA på rätt sätt.
DNA-polymeraset måste vara
- noggrant - kallas även fidelitet
- annars mutationer och kanske sjukdomar
- effektivt - kallas processivitet
- måste fungera på väldigt långa sträcker, vara väldigt noggrant och en hög processivitet, långa sträcker utan att falla av
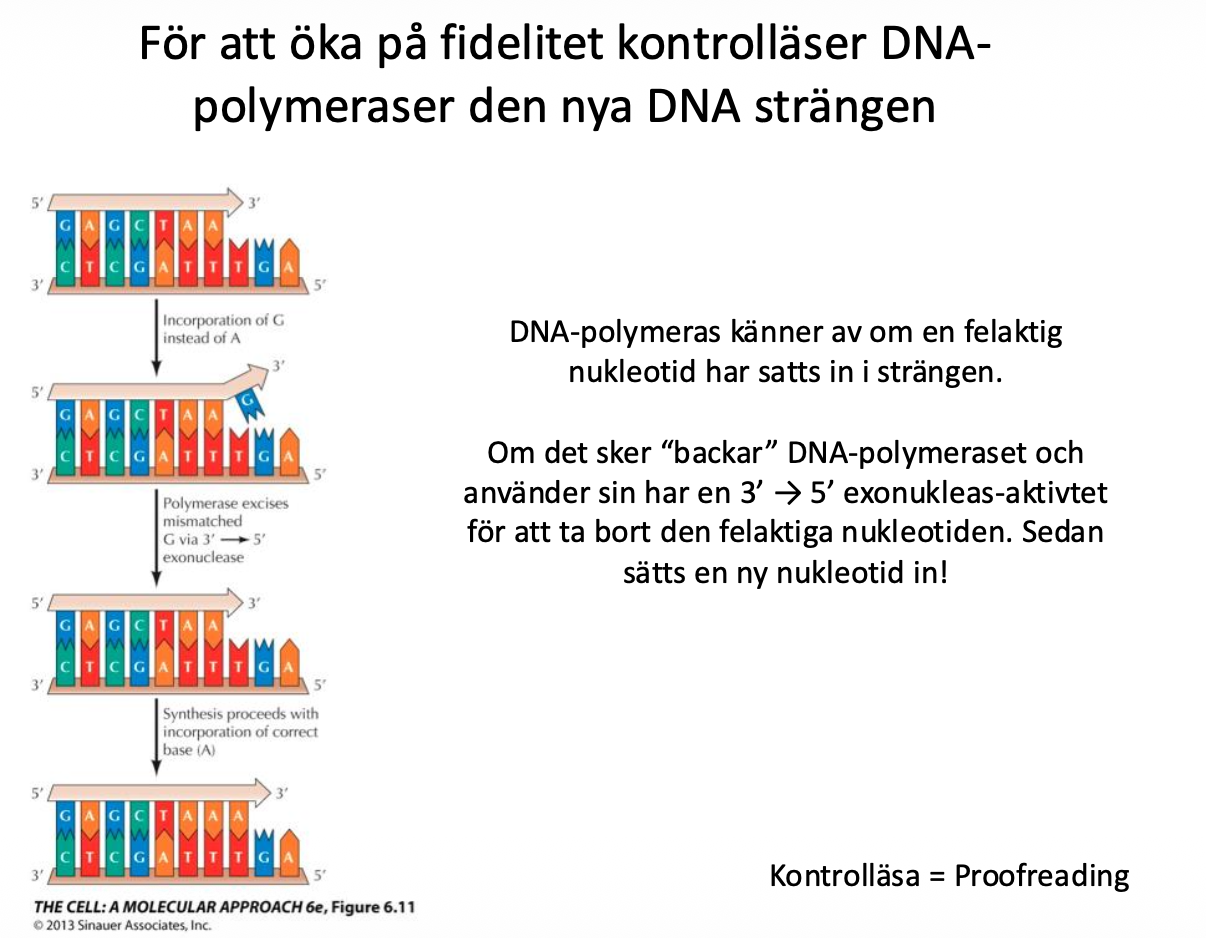
Ibland blir det fel, kommer in en felaktig nukleotid
Det finns en möjlighet för DNA-polymeraset att fixa det, det har inte bara
- att röra sig framåt
- men har också möjligheten att backa
- exonukleoasaktivtet (ta bort ifrån änden)
Förmågan att backa kallas proofreading
SLIDE endonukleaser/exonukleaser saknas
endonukleas
- ta bort i mitten exonukleas kan delas upp i två grupper
- ta bort i en ände
- 5’
- 3’
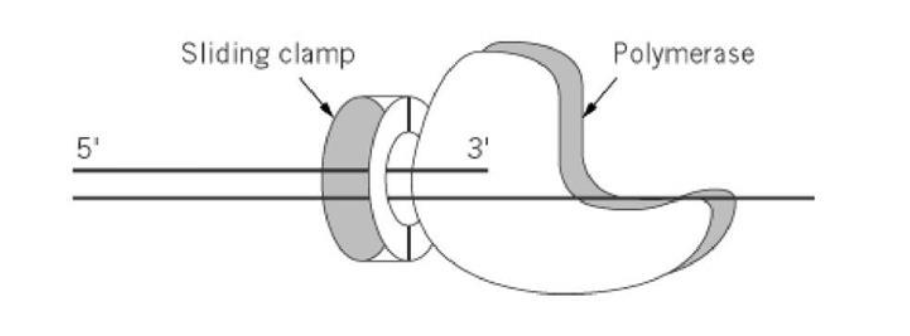
Måste vara processiva (effektiva!) Miljoner, miljarder baspar som måste replikeras Måste köra under en lång tid
Processivitet = förmågan att koppla på många nukleotider till den växande DNA-strängen, utan att polymeraset lossnar från mallsträngen.
sliding camp - klämma
- sätter sig fast i änden
- fungerar som ett ankar som håller kvar DNA
- ökar effektivtet 50ggr för att DNA-polymeraset inte lossnar
- utan: 10-20 nt/s
- med: 500-1000nt/s
Problem vid DNA-replikation (2) DNA-polymeraser kan inte starta DNA replikation de novo. De behöver en primer.
de novo = från ingenting
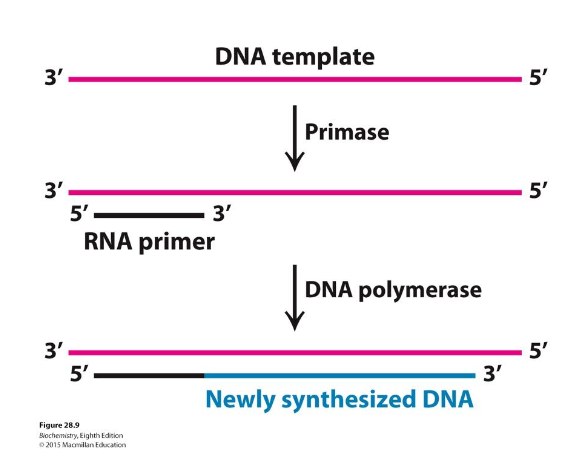 För att börja, behöver vi en liten startpunkt som är en RNA primer.
För att börja, behöver vi en liten startpunkt som är en RNA primer.
RNA-polymeras kan startas de novo DNA-polymeras behöver en RNA-primer
Finns ett specialiserat DNA-polymeras som bara gör väldigt korta strängar, behöver bara en liten snutt, som det vanliga DNA-polymeraset kan starta och köra igång
- det kallas för DNA-primas
I våra celler sitter DNA-polymeras tillsammans med DNA-primas (kommer senare)
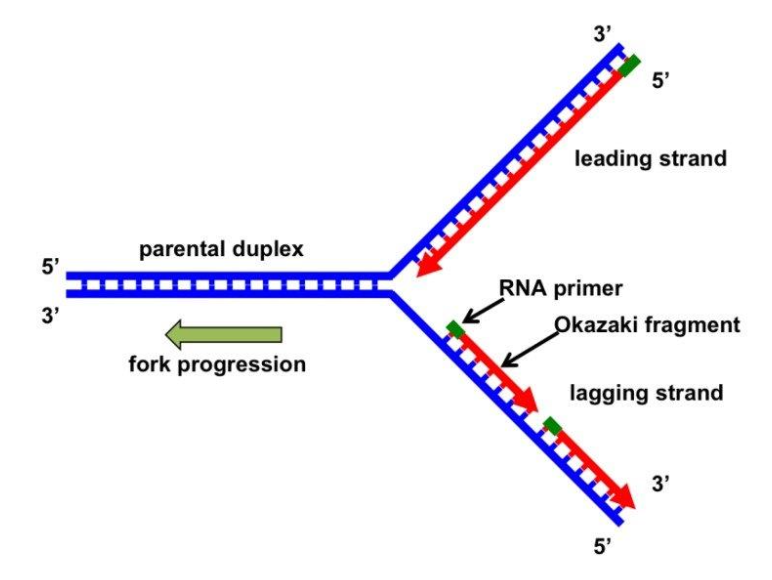
leading strand: primers behövs bara en primer lagging strand: en primer per fragment FRÅGA: hur lång är en fragment FRÅGA: varför kan man inte bygga bakvänt?
- har med fosfatbryggan, naturen har valt att göra det FRÅGA: plockas primern bort?
- vi vill inte ha RNA i DNA
- vi vill inte ha en lucka
Problem vid DNA-replikation (3) På lagging-strängen finns en RNA-primer i början av varje Okazaki-fragment! Vi vill inte ha sträckor av RNA i DNA-molekylen!
Energin kommer med den inkommande nukleotiden det bestämmer att reparation måste gå i rätt riktning trifosfater är lite instabila, de kan gå sönder, sitter de på DNA-kedjan kan det bli katastrof.
okazaki-fragement kan variera i längd, när de nått en viss längd så lossnar maskineriet
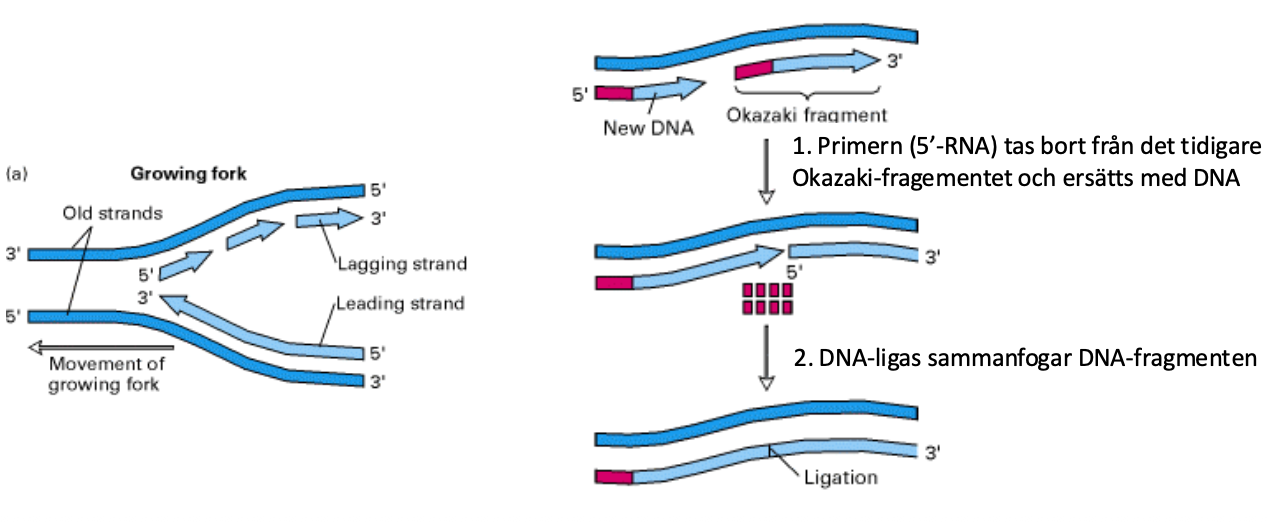
VIll ta bort - fragement maturation
- steg 1 RNA-primer
- steg 2 brytpunkten, DNA-fragmenten måste sitta ihop ordentligt
- försluter ryggraden så den blir hel
- kallas ligering - klistrar ihop fragmenten
- får inte finnas brott (nicks)
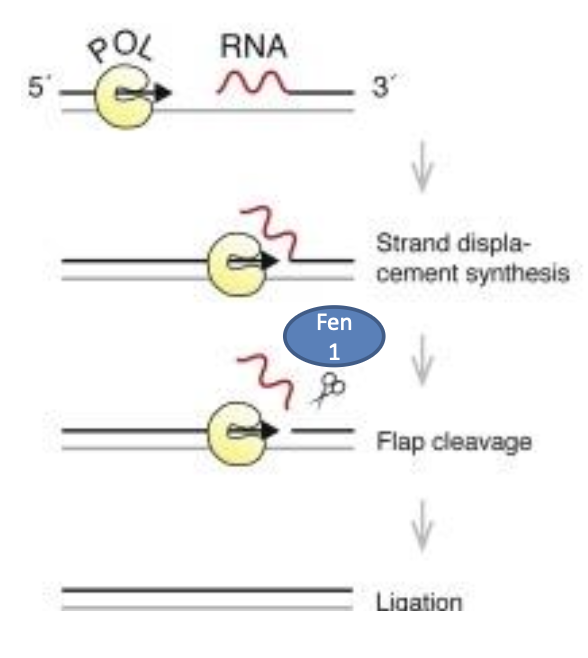
Steg 1
Från vänster närmare sig det nya fragmentet DNA polymerases stannar inte, det krashar in i RNAt, när det händer så släpper det från DNA, de sitter inte längre hybridiserat. Bildar en lite flärp, flap. Som petar ut det här RNA, medan DNA finns kvar på strängen
Finns ett speciellt endonukleas som känner igen detta flappar, kallas Flap Endonukleass 1 eller FEN1 som kan komma in och klyva. Sen finns det bara en litet nick kvar, allt RNA är borta. Det är eukaryota celler när vi ta bort
Steg 2
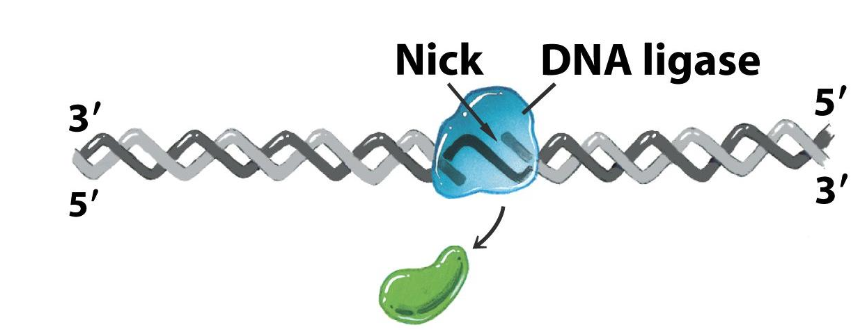 Använder energi från ATP för att laga nicken, det är DNA-ligas som lagar.
Själva nicken är avsaknaden av en fosfodiesterbrygga, alla nukleotider finns men en brygga saknas.
Använder energi från ATP för att laga nicken, det är DNA-ligas som lagar.
Själva nicken är avsaknaden av en fosfodiesterbrygga, alla nukleotider finns men en brygga saknas.
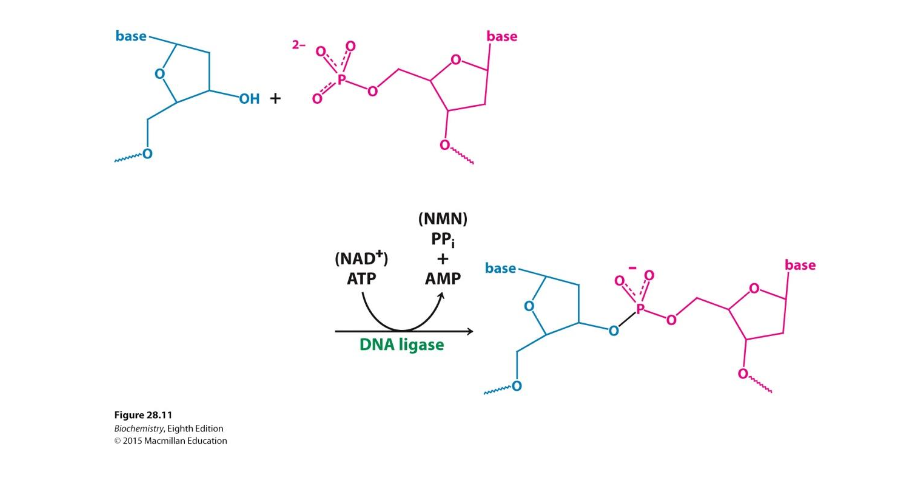 Använder ATP som en ko-faktor som kan klistras ihop.
Använder ATP som en ko-faktor som kan klistras ihop.
Problem vid DNA-replikation (4)
DNA är dubbelsträngat! Hur kan vi dela på strängarna så att DNA replikation kan ske?
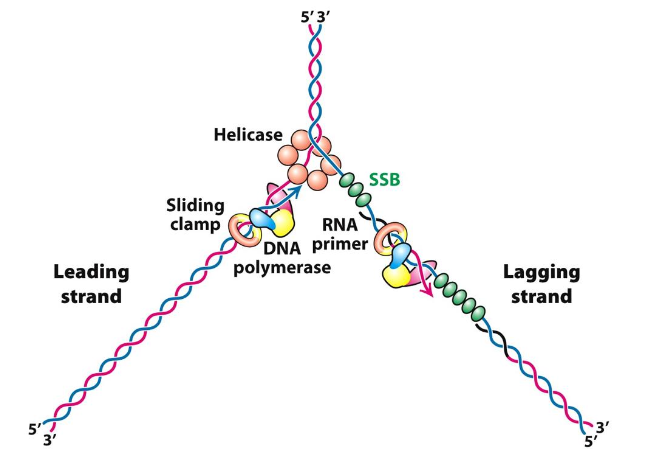
Helikas är en grupp av enzym:
- en förmåga att dela DNA
- finns olika beroende på process
I DNA-replikationen sitter helikaset längst fram, det är det som gör att gaffeln kan röra sig.
- det binder till en av strängarna, fungerar som en kil
- klättrar på en sträng, så pressar det isär DNA som finns framför
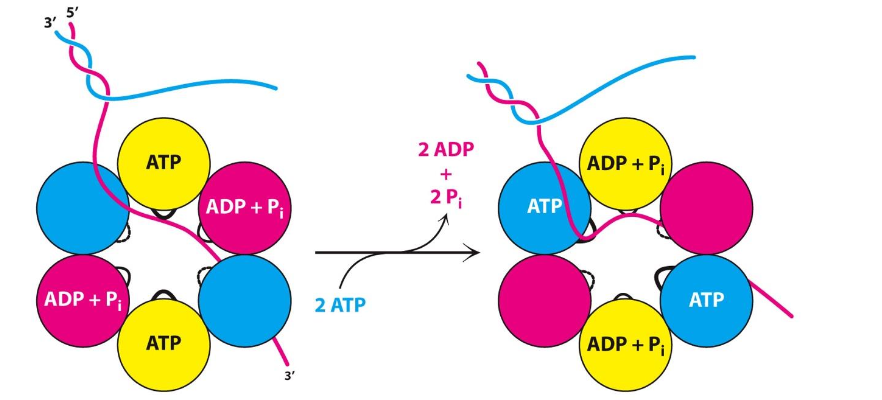
- Består av 6 st identiska subenheter
- Den bildar runt DNA
- Har förmåga att klättra som en repklättrare
- Hydroliserar ATP använder energi från det
- den rör sig upp för det här och så växer gaffel, så går den längre och längre uppför
- klättrar utmed
- snurrar runt lite här
- alla olika subenheter kan hydrolisera ATP, det blir som en rörelse uppåt
På leadning strand sitter DNA-pol en liten bit efter För lagging strand är det lite mer komplicerat, måste finns upp till 200 nt lång sträcka innan man syntesiserar, måste skydda DNA i den biten, känsligt för omgivningen, mycket som reagera, som går in och basparar med sig själv Måste stabilisera det enkelsträngade DNA så inte det sker
Kallar de för enkelsträngadeproteiner
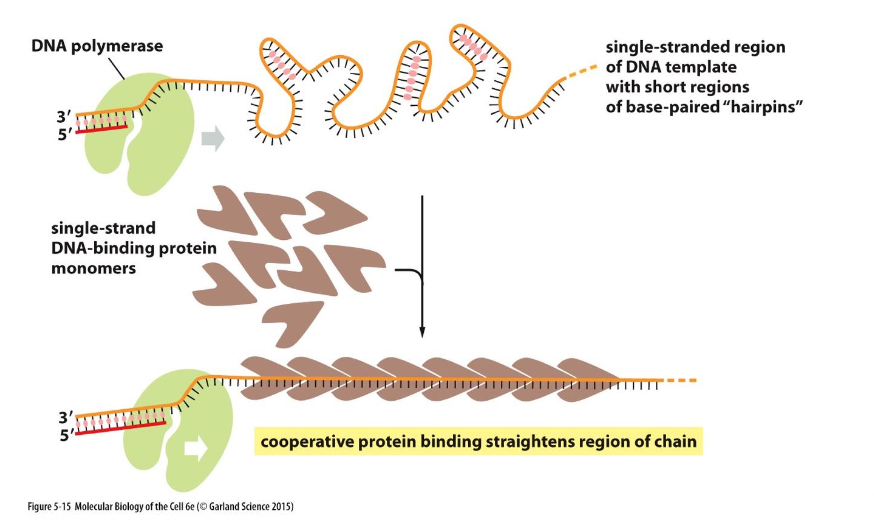
Allt det här stimulerar DNA-replikation, för att det ska kunna ske effekivt I de flesta celler kallar det single-cell-binding protein
Replication Protein A kallas det i våra celler RPA, spelar en viktig roll här.
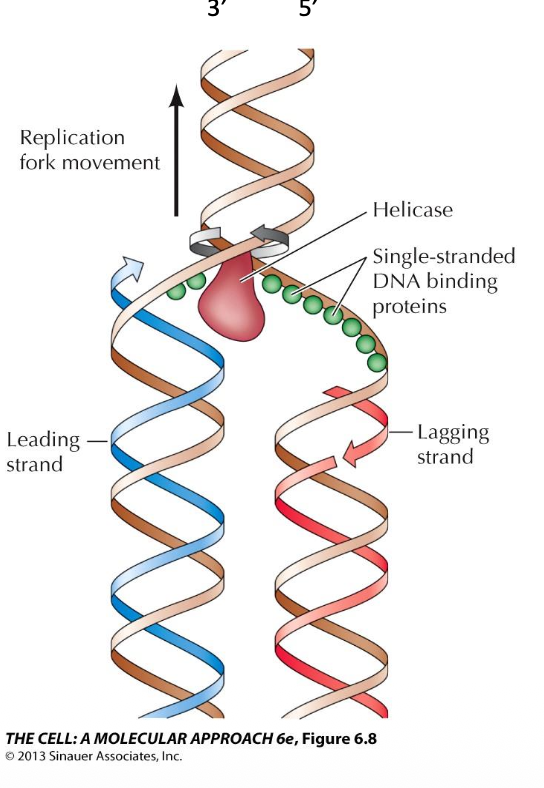 Proteinerna fungerar tillsammans som ett maskineri, de viktig att alla är på plats tillsammans
Ofta bildar de interaktioner mellan varandra
Helikaset känner av att det finns ett single-stranding DNA binding, de stimulerar varandra så de vågar röra sig framåt, de blir mer effektivt.
Om man återskapar det här i ett provrör, om man lägger till ett single-stranding DNA.
Proteinerna fungerar tillsammans som ett maskineri, de viktig att alla är på plats tillsammans
Ofta bildar de interaktioner mellan varandra
Helikaset känner av att det finns ett single-stranding DNA binding, de stimulerar varandra så de vågar röra sig framåt, de blir mer effektivt.
Om man återskapar det här i ett provrör, om man lägger till ett single-stranding DNA.
www.youtube.com/watch?=l9ArIJWYZ
Problem vid DNA-replikation (5)
När man tvingar isär dubbelhelixen så skapas topologiska problem.
Varför och hur löser cellen detta?
När vi gör en DNA-replikation kan snörerna svängarna/helixarna, har ingenstans att ta vägen, de kommer bli massa spänningar i det här DNA. Tänk skosnöre som är tvinnat. Man kan inte förvänta sig att det ska lösa sig självt.
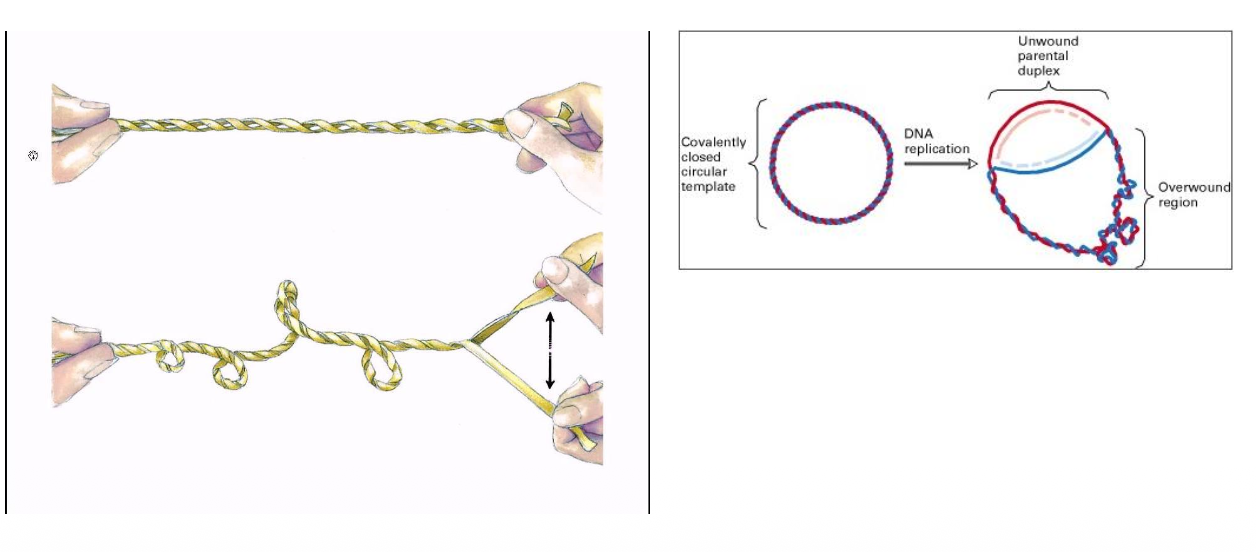
Det blir massa spänningar och jobbigt, massa konstiga sekundärstrukturer. Som heter supercoils
Det blir mkt motstånd, måste bli av med spänningarna, behöver någonting framför för att slappna av DNA:t
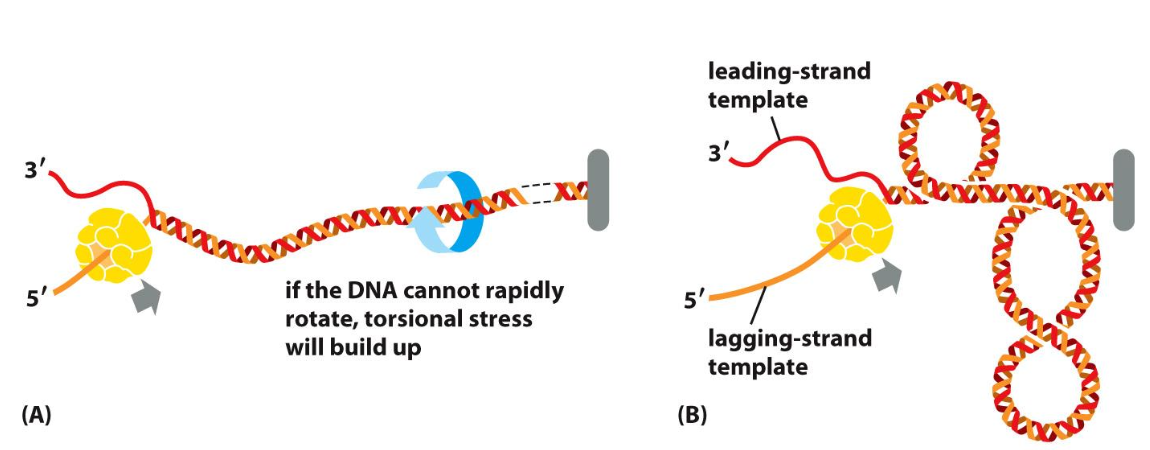
Går inte att snurra DNA-molekylerna, de blir fixerade och tar man bara helikaset. Ju mer man kör ju mer spänningar, konstiga strukturer, stannar replikationen. Får detta att slappna av.
Linking DNA
- Har ungefär 10.4 bp/varv. Fin avslappnad och optimerad DNA-helix.
- 160bp lång, får plats med 25 varv
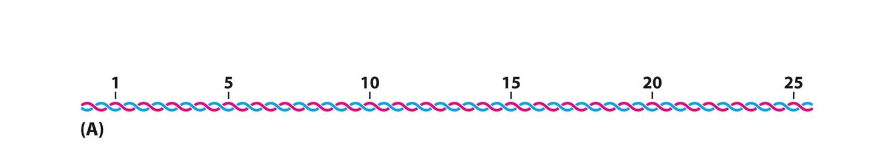
Man kan slå ihop de, så blir de så fint här:
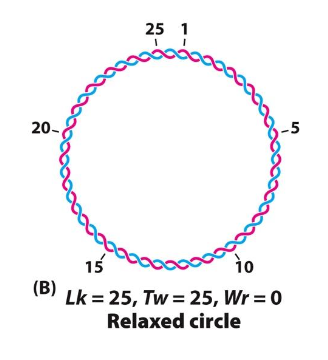
Men vad händer om man introducerar 5 extra varv, då bygger vi upp spänningar i molekylen. Linking Number (Lk) är antal varv, det ska motsvara det optimera och helst vara 25 varv för 160 bp
- vad händer om det är mer eller mindre
- då förstör man DNA, det skapar topologisk stress
Stress = fler antal varv än DNA strängarna
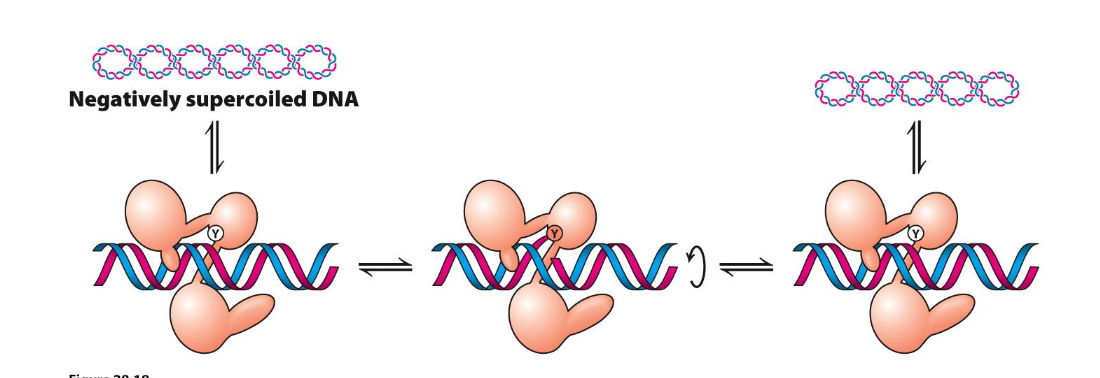
Topoismeraser är de som tar bort spänningarna i DNA.
Det är specilla enzymer. topo=läge iso=samma meris=del -as=enzym
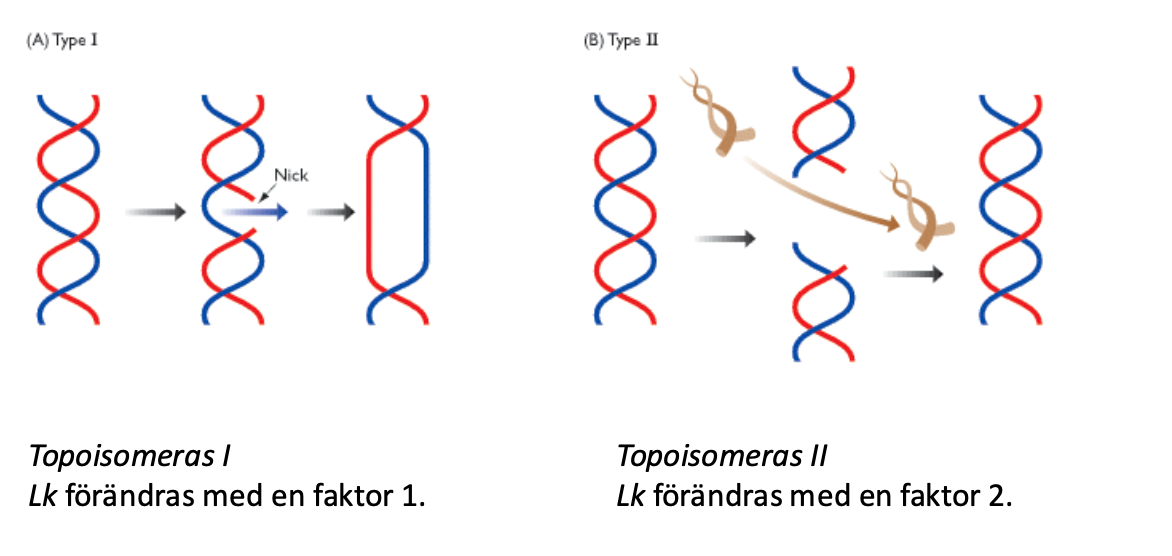
Typ I: - tar bort supercoils, öka linking number är mer korrekt - kräver inte ATP Typ II: - Tar bort men kan också skapa, kräver ATP
Typ 1
Klyver ena strängen och tar bort ett varv i taget
- ändrar med en faktor av 1
- använder energi i spänningen
 Typ II
Typ II
- den klyver båda strängarna och låta en sträng
- låta en sträng komma igenom, den gör dubbelsträngat brott
- två varv i taget
- ändrar med en faktor av 2
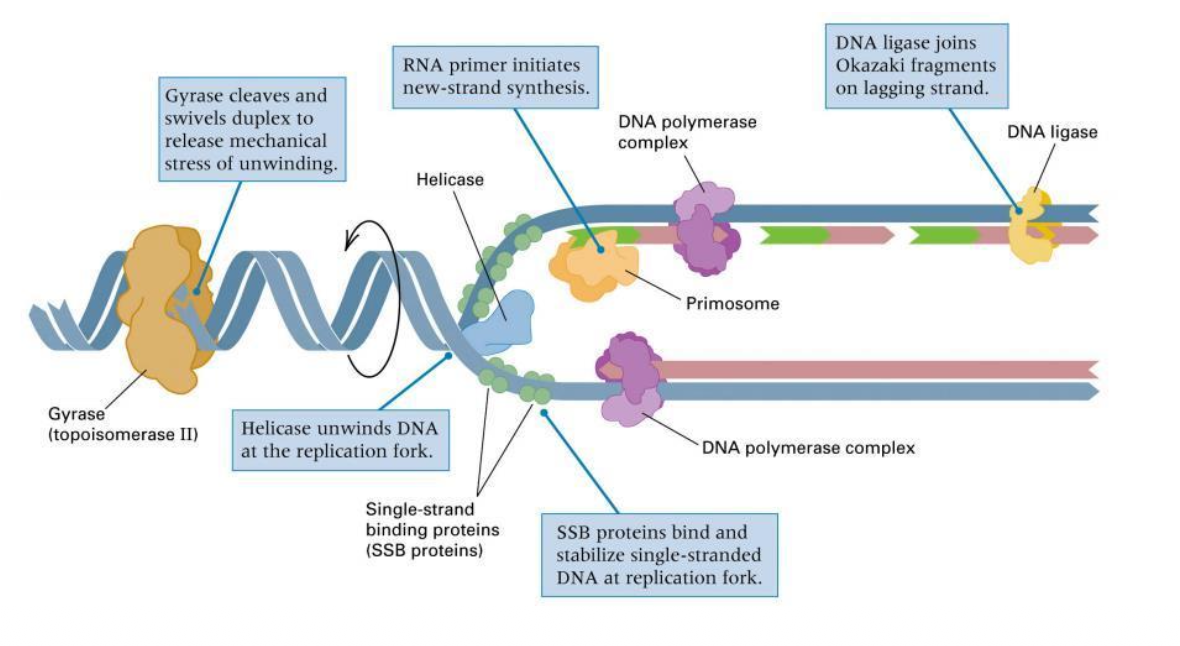
Framför replikationsgaffeln sitter det topoisomeras II framför replikationsgaffeln och tar bort positiva supercoils
I bakterier kallas topoisomeras typ II ofta för Gyras. Gyras-hämmare är en viktig grupp av antibiotika! Tex. Ciprofloxacin för urinvägsinfektioner de är bredspektrumantibiotika eftersom det är mot många